
文章信息
论文标题:Dynamics of the human and viral m6A RNA methylomes during HIV-1 infection of T cells.
刊登日期:2016年02月
发表杂志:Nature Microbiology
影响因子:14.174
研究机构:加州大学圣迭戈医学院Tariq M.Rana团队
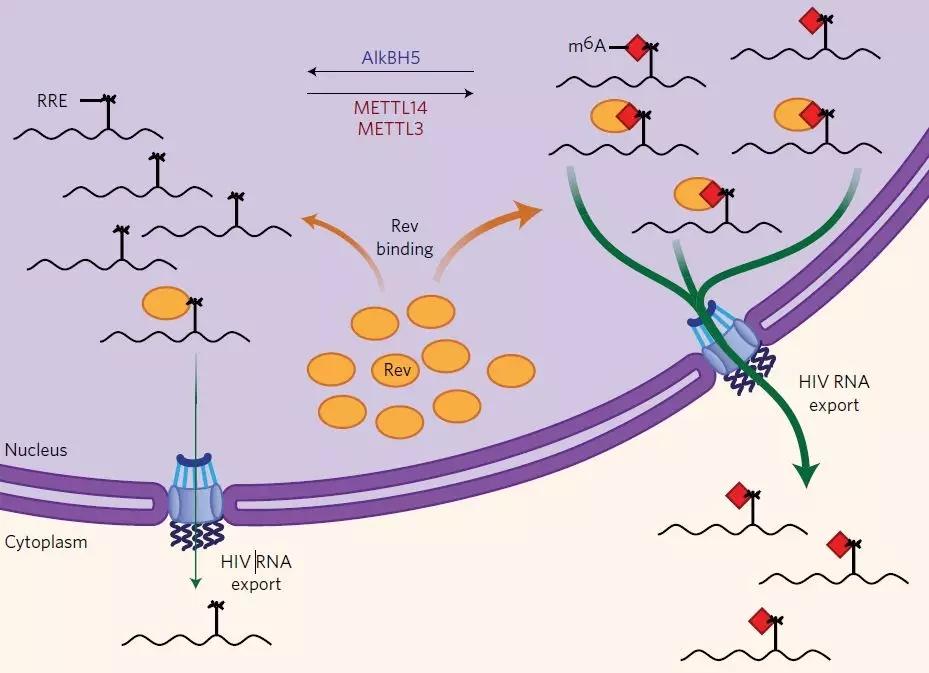
m6A是真核生物mRNA上最普遍的一种修饰方式之一。但是m6A在免疫系统中的作用或其在宿主-病原微生物互作方面的研究,相关论文依然不多。来自加州大学圣迭戈分校的Rana团队研究了HIV侵染最常见的一种免疫细胞——CD4 T细胞。作者首先针对细胞受到HIV-1侵染后MeRIP-seq的测序结果进行分析,然后作者针对病毒和宿主关于RNA甲基化的互作机制进行了深入研究。结果表明,HIV-1感染T细胞后触发病毒和T细胞mRNA上的m6A整体修饰水平大大增加。在HIV-1 mRNA中,作者在编码区和非编码区等区域通过MeRIP-seq鉴定了14个m6A peak。此外,作者还从感染HIV的T细胞的MeRIP-seq测序结果中鉴定出56个宿主基因在m6A水平上发生差异,这些基因对于病毒基因的复制至关重要。关于病毒上m6A修饰与病毒复制本身是否存在相关性,作者通过对宿主体内甲基化转移酶及去甲基化酶的knockdown来进行验证,m6A修饰能够增强病毒本身的复制和出核能力。最后,HIV-1的Rev蛋白与RRE上RNA茎环IIB区能够互作是由于A7877和A7883上携带有m6A修饰。对A7877突变后并不影响HIV-1的生物学功能,但是A7883突变后会严重影响病毒的复制及出核速度。
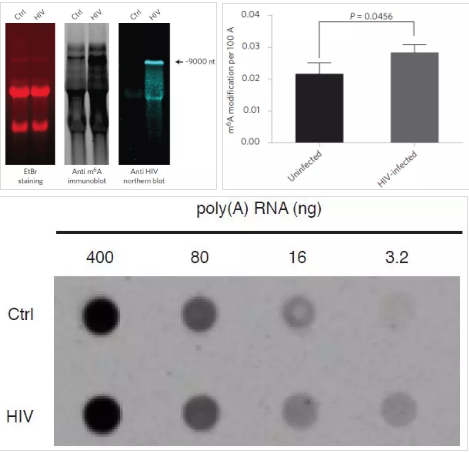
为了证实HIV-1的RNA是否在病毒侵染的过程中被修饰,作者在病毒处于活跃复制期从HIV-1侵染3天后的MT4 CD4+ T细胞(即LAI克隆)中提取了total RNA。使用免疫印迹法分析total RNA整体的m6A水平,并用LC-MS/MS进行了进一步验证。感染HIV-1的T细胞和未感染HIV-1的T细胞相比,m6A整体水平升高了30%。Total RNA整体的甲基化水平都有所增加表明了病毒对细胞整体RNA的甲基化修饰水平具有广泛的影响,作者同时对核糖体RNA(rRNA)也进行了免疫印迹实验来证实这个观点。这些结果均表明T细胞在受到HIV-1侵染后病毒以及宿主整体的m6A水平都有所上升。
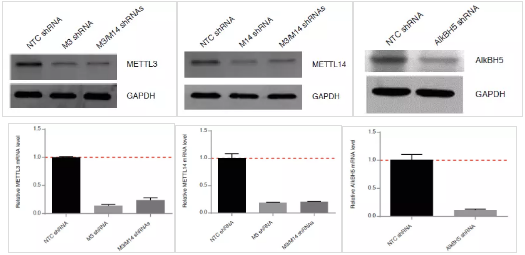
接下来作者需要确认m6A的丰度发生变化是否会改变HIV-1的复制效率。首先使用shRNA干扰甲基化转移酶METTL3、METTL14和去甲基化酶ALKBH5,并通过RT-qPCR和WB进行了验证。作者选择对ALKBH5进行敲低,因为这个基因本身在T细胞里表达水平较高。
T细胞在感染HIV-1病毒3天后,通过RT-qPCR分析GP129的mRNA转录水平和P24细胞提取物的大小来验证病毒的复制效率。与NC对照相比,METTL3和METTL14单独敲低后能够显著减弱病毒的复制能力,当2个甲基化转移酶同时被敲低时,病毒复制能力加倍减弱。相反ALKBH5被敲低后由于病毒m6A水平上升而导致病毒复制能力有所增加。
综上所述,METTL3、METTL14、ALKBH5的表达能够直接影响HIV-1复制。
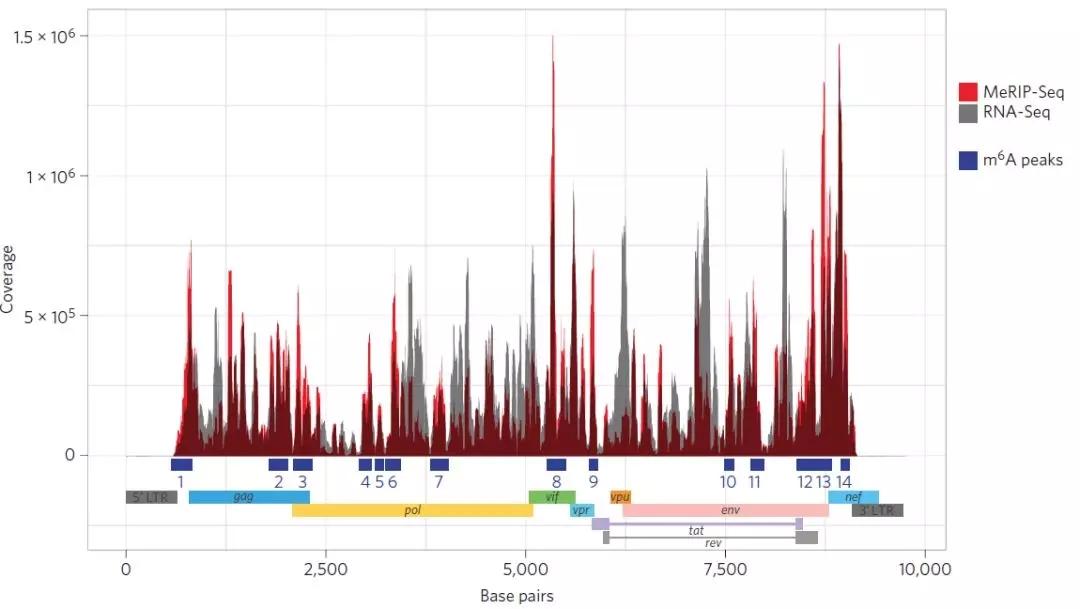
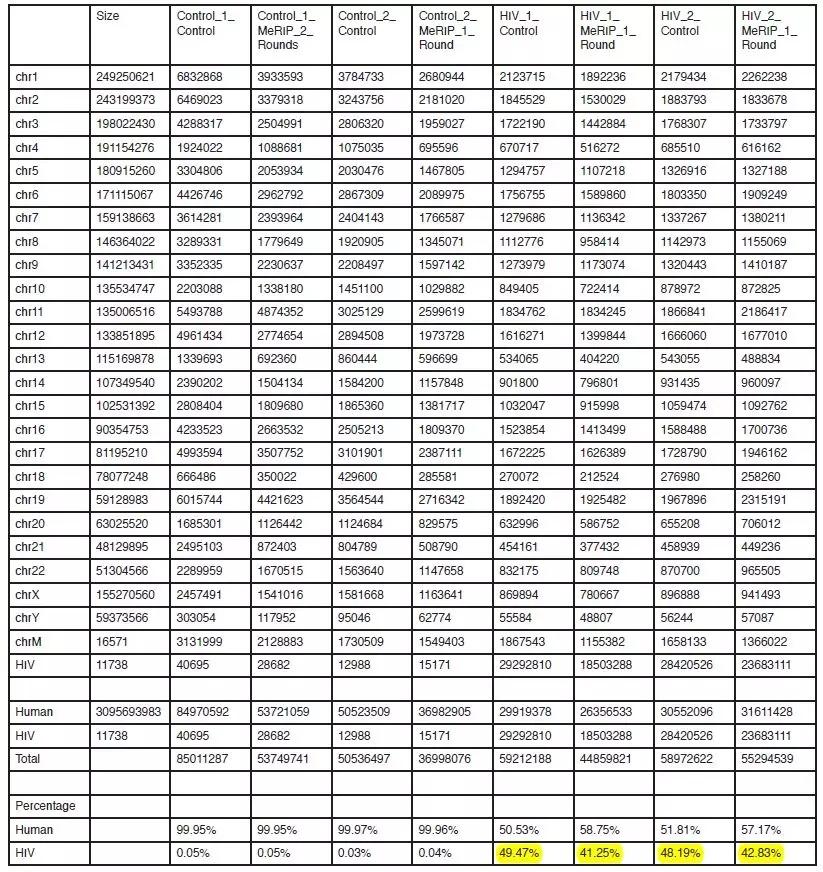
接下来,作者通过MeRIP-seq研究HIV-1感染T细胞后究竟病毒以及宿主本身的哪些基因或区域发生m6A甲基化差异。结果MeRIP-seq中INPUT数据和IP数据中分别有49%和42%的reads比对到了HIV的基因组上。共在HIV基因组上鉴定出14个peak峰,分别位于编码区、非编码区以及剪切连接点Splicing junctions D1区及剪切调控区。每个峰所在的区域都代表着m6A修饰具有潜在的生物学功能。这些被鉴定的peak区域表明在HIV整个基因组中广泛存在m6A修饰。
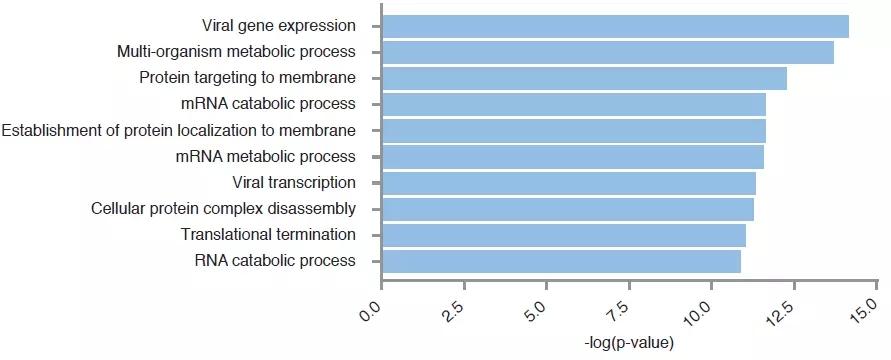
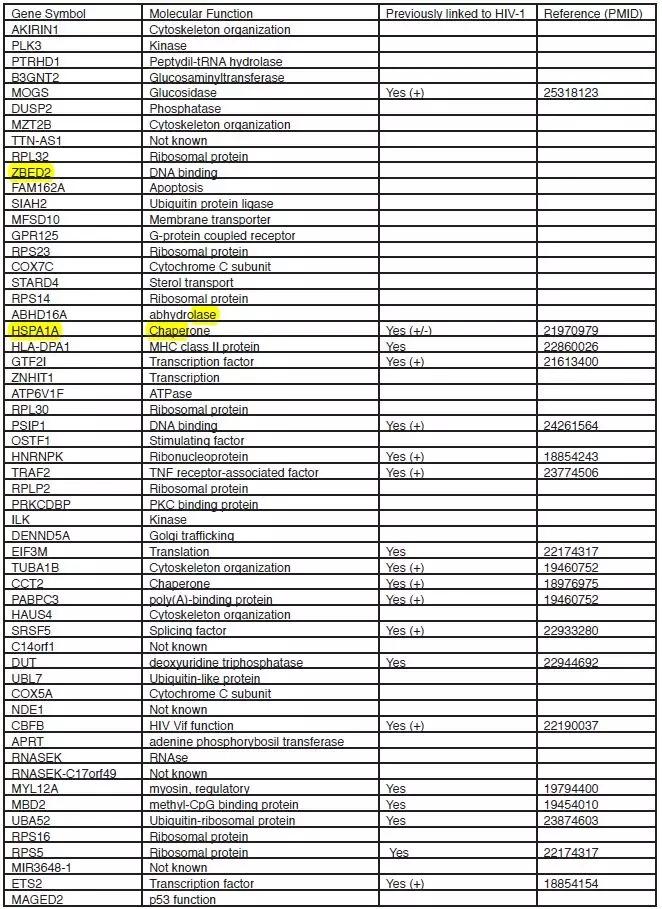
下一步,作者通过MeRIP-seq对HIV-1感染后的T细胞进行研究。针对m6A甲基化水平发生差异的基因进行功能富集分析后,进一步鉴定出56个与病毒基因复制相关的特异性差异基因。事实上,在这56个基因中,有19个与HIV-1复制相关,并且在某些情况下,这些宿主蛋白能够直接与病毒的某些成分相互作用。值得注意的是,这些基因大多数会编码具有proviral功能的蛋白质。敲除MOPS,PSIP1(LeDGF)、HNRNPK、TRAF2、TUBA1B、CCT2、PABPC3、CBFB和ETS2能够对不同的病毒复制步骤有负调节作用;病毒侵染宿主后会激活GTF2I、MYL12A和UBA52的表达。HLA-DPA2、EIF3M、DUT、MdD2和RPS5蛋白能够与病毒蛋白互作,而HSPA1A和SRSF5过表达会促进病毒的复制。
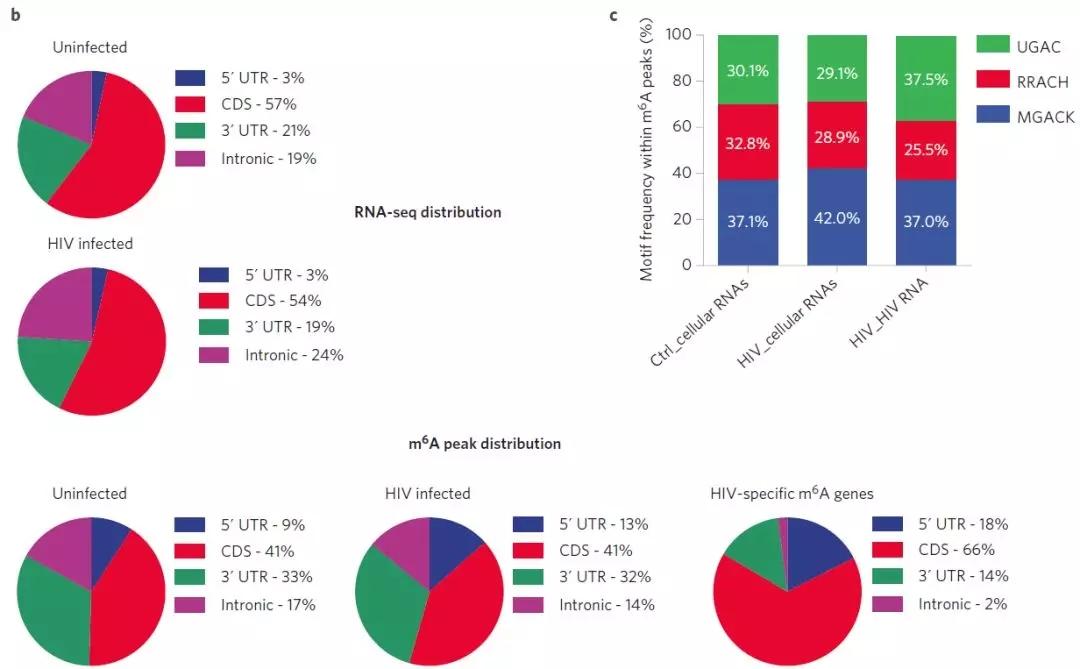
关于m6A如何调控RNA转录本的稳定性、剪切及翻译,目前这些机制研究尚未清楚。作者接下来比较了56个特异性基因的peak特征后发现了一些显著差异。未感染T细胞结果中,peak位于5’UTR、CDS、3’UTR和内含子区域的reads比例分别是17.5%、66%、14.4%和2.1%,而HIV-1感染的T细胞比例则是13.4%、41%、31.6%和14%。因此对于HIV-1感染的T细胞而言,m6A更多是富集在5’ UTR和CDS区,而3’ UTR和内含子区则m6A丰度较低。这些结果表明m6A修饰可能针对mRNA的剪切与翻译而不是维持mRNA的稳定性。此外,m6A修饰更多情况下存在于成熟的mRNA上而不是非成熟mRNA(有时候可能是pre-mRNA即mRNA前体)上,例如HIV-1 RNA的内含子区的m6A峰与细胞中整体mRNA相比有所降低。
作者对HIV病毒RNA以及宿主RNA进行motif分析,并且进一步研究这些motif是否在HIV-1侵染后发生了改变。在未发生HIV-1感染的T细胞中,MGACK (A/C-GAC-G/U)、RRACH (G/A-G/A-AC-A/C/U)和UGAC motif的比例分别是37%、33%和30%。HIV-1感染后MGACK motif的比例上升到37%~42%,这表明病毒感染期间motif的比例发生较大的变化。在HIV-1病毒RNA中,UGAC和MGACK基本比例基本都是37%,但UGAC比例比宿主细胞升高了7%。
虽然病毒感染宿主后motif比例发生变化的机制尚未知晓,但是作者仍然做了一些推测。例如甲基转移酶蛋白复合物在感染HIV-1之后催化速率发生变化,对motif偏好的优先顺序发生改变。相反,HIV-1侵染T细胞后导致去甲基化酶发生改变降低了MGACK的识别,但不能降低其他motif的识别。又或者这些motif通过隔离或二级结构的改变而发生变化。
在未来几年内,作者认为解析HIV-1感染T细胞后甲基化转移酶蛋白复合结构,无疑将会使人们从更深的层次去理解哺乳动物细胞以及病毒RNA甲基化与病毒入侵宿主之间的关系。
考虑到m6A修饰可能会改变RNA的功能并促进HIV-1复制,作者推测m6A修饰可能会改变RNA的二级结构或三级结构,从而影响其与蛋白互作的能力。
RRE是HIV-1 env基因编码一种RNA元件。翻译后,Rev蛋白会重新返回细胞核中,在RRE中组装成出核蛋白复合物,帮助病毒RNA进入胞浆。这是病毒复制的一个重要的步骤。
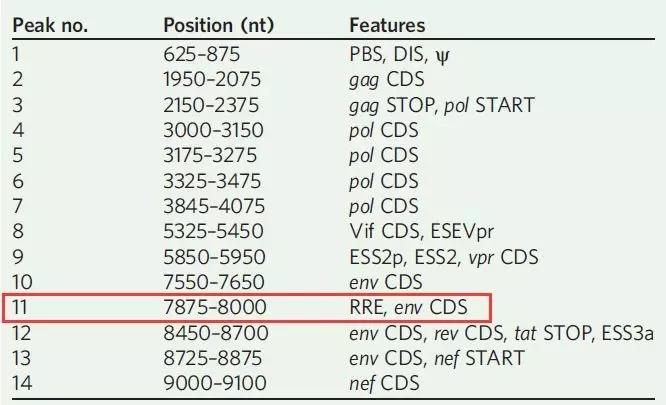
有趣的是,RRE RNA的茎IIB区域位于HIV-1基因组上第11个peak,并且含有大量的Rev高亲和结合位点。RRE的二级茎环结构是与Rev蛋白相结合的关键所在,并且与病毒RNA出核相关。Rev与IIB茎环结构相“捆绑”的溶液结构表明Rev结合在RRE一个独特的结合口袋中。因此,茎环上的A7877和A7883发生m6A修饰可能会影响RRE与Rev蛋白的结合能力,从而改变了RNA-蛋白复合物的功能。
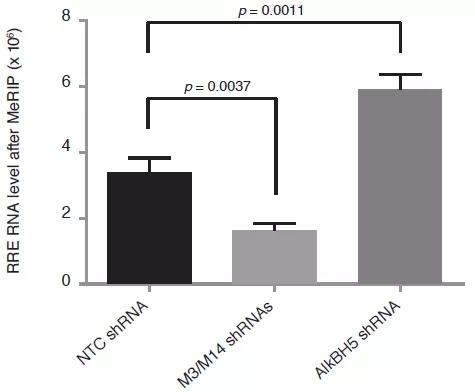
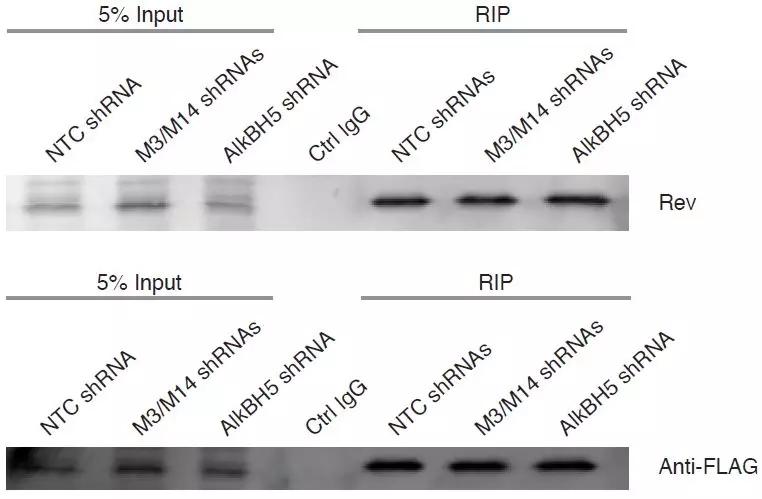
为了进一步探究Rev蛋白与RRE互作的分子机制,作者在T细胞中敲低METTL3、METTL14和ALKBH5后使用MeRIP-qPCR对RRE进了验证后发现,甲基化转移酶和去甲基化酶确实对RRE的甲基化水平发生了改变。沉默METTL3和METTL14后降低了RRE的m6A水平,抑制ALKBH5后RRE的m6A水平上升。
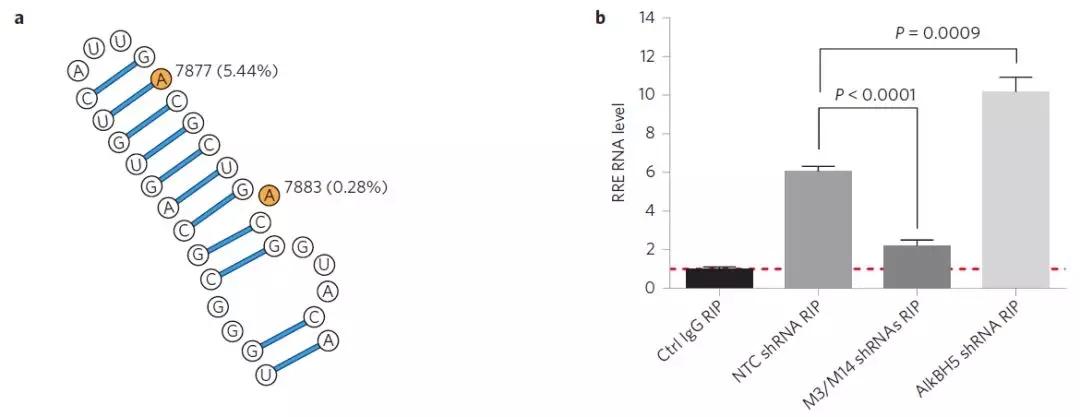
为了确定RRE上m6A水平发生改变后是否会影响其与Rev结合的亲和力,作者在293细胞中瞬时表达了HIV-1。对METTL3或METTL14沉默之后,采用Rev抗体的RIP-qPCR证实Rev-RRE互作能力降低。而ALKBH5沉默后,结果相反,即Rev-RRE互作能力增强。
所以以上结果已在体内实验(in vivo)证实RRE上m6A修饰能够促进RRE与Rev互作。Rev CO-IP的分析表明,这些效应不是由于表达不同shRNA的细胞之间的Rev CO-IP效率的差异所导致的。
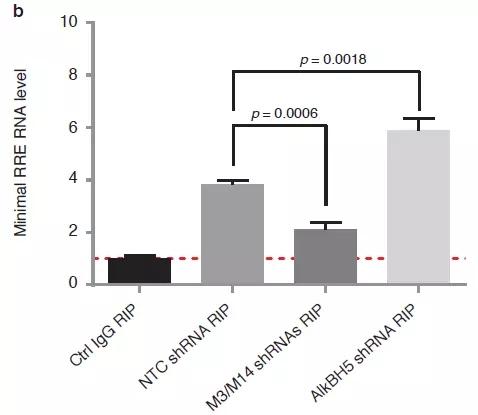
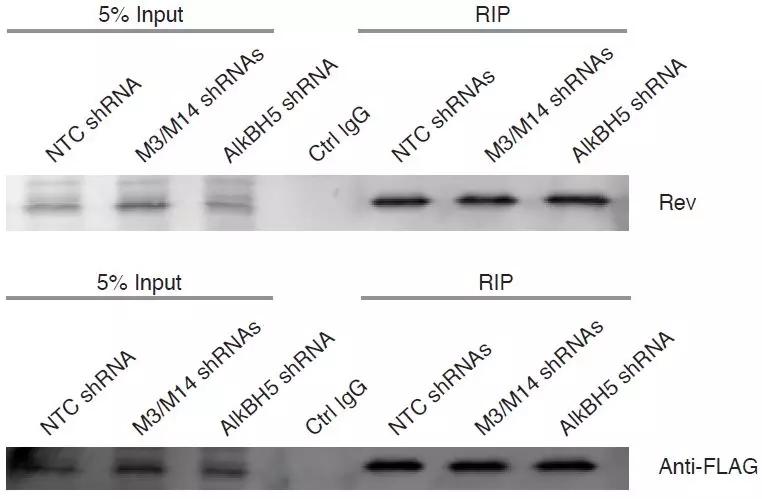
由于在293细胞中表达的是HIV-1的全长,可能HIV-1的其他蛋白也会影响Rev-RRE互作。为了解答这个问题,作者表达了RRE茎环结构最小的66nt序列长度并携带FLAG标记,其结果与HIV-1全长结果一致。所以这些证据均强有力证实RRE茎环IIB区上A7877和A7883发生m6A修饰后能够影响其与Rev蛋白结合。
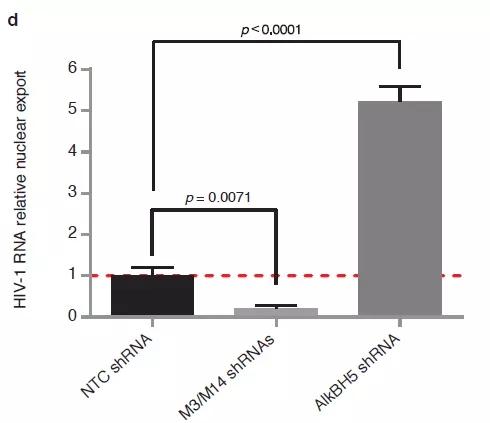
Rev-RRE互作及其在病毒RNA出核中的功能属于HIV-1复制循环周期中十分重要的一环。下一步,作者将要验证RRE上m6A修饰发生改变后能够改变病毒RNA的出核能力。在293细胞中敲低METTL3、METTL14和ALKBH5后感染HIV-1,并对Rev-RRE互作以及细胞中部位分布进行定量分析。结果表明敲低METTL3和METTL14后,病毒RNA出核能力显著降低,而敲低ALKBH5则会加剧病毒RNA出核能力。
为了在RRE的发夹结构中确定究竟是哪个A碱基发生了m6A修饰,作者用AMV RT酶和Tth聚合酶这种特殊的方法,对A7877和A7883上m6A修饰序列特异性互补单链DNA探针进行酶扩展分析。这种方法已由斯坦福大学化学系的Kool教授发表在2013年美国化学协会期刊(Journal of the American Chemical Society)上。最后A7877和A7883上的m6A修饰通过ssDNA探针以及一种特殊的酶扩增得到验证。
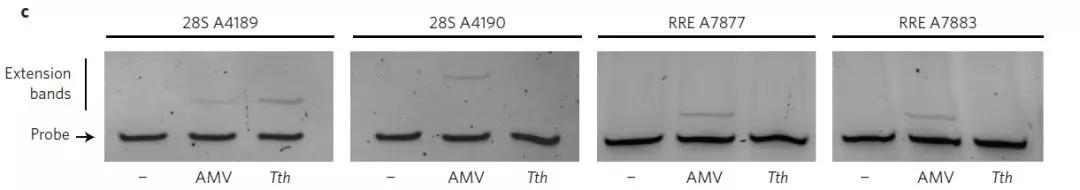
如上图所示,Tth在A7877和A7883这2个位点都不能对探针进行延伸。在对照实验中,Tth pol扩展oligos为A4189但并没有扩展到A4190(m6A修饰位点)。这个结果与Kool教授的结果一致。
总之这些结果均表明,腺嘌呤A及RRE发夹结构均存在m6A修饰且影响Rev结合能力至少存在2个机制:特定Rev氨基酸残基与甲基化集团互作或诱导RRE折叠导致其结构发生改变后影响Rev识别能力。当然这些机制仍然需要更多的研究来证实。
小贴士
虽然近几年人们已经在动植物中成功地鉴定了许多含有m6A修饰的转录本(mRNA、lncRNA和circRNA等),但是很难确定mRNA以及部分lncRNA上m6A修饰的确切位置,更多的时候只能进行费用昂贵的方法如m6A-CLIP-seq。斯坦福大学化学系的Eirc Kool教授已经从嗜热菌Thermus thermophilus中鉴定出具有逆转录酶活性的聚合酶。这种酶针对未发生m6A修饰的腺嘌呤互补配对的胸腺嘧啶具有高达18倍的选择亲和性。所以通过分析断裂的电泳条带,这种酶能够用于定位和量化m6A在RNA上的具体位置。所以某种意义上,部分老师可以选择不使用CLIP的方法也能对RNA上具体的m6A修饰的位置进行精确定位(低通量验证)。

参考文献:Harcourt et al. Identification of a selective polymerase enables detection of N(6)-methyladenosine in RNA. J Am Chem Soc 135.51(2013):19079-19082.(pubmed:24328136)
RRE上A7883甲基化影响HIV-1复制和出核
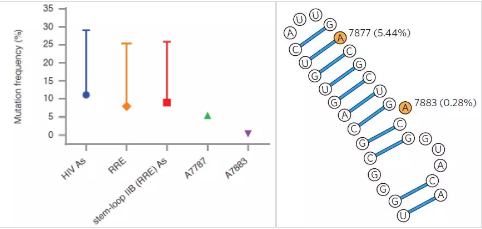
为了证实RRE上m6A修饰能够“赋予”HIV-1进化上的优势,作者针对数据库中2501条HIV-1序列中A7877和A7883的突变频率进行了生信分析。其中A7877和A7883上5.44%和0.28%的突变频率远远低于RRE的IIB茎环上其他区域。这就表明RRE上A7877和A7883这2个位点在进化上较为保守且有利于病毒存活,有趣的是A7883位于RRE茎环IIB区与Rev高亲和性的结合结构域中。
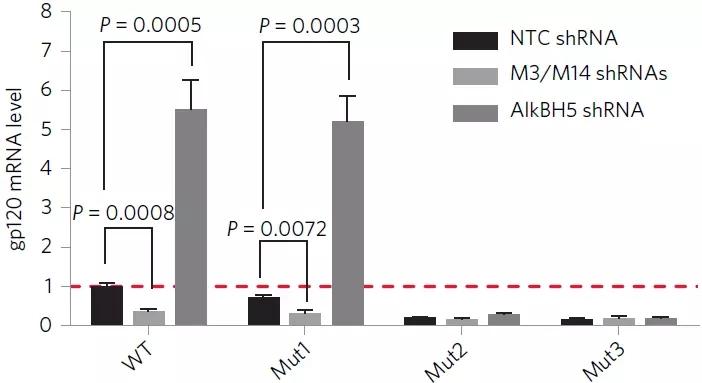
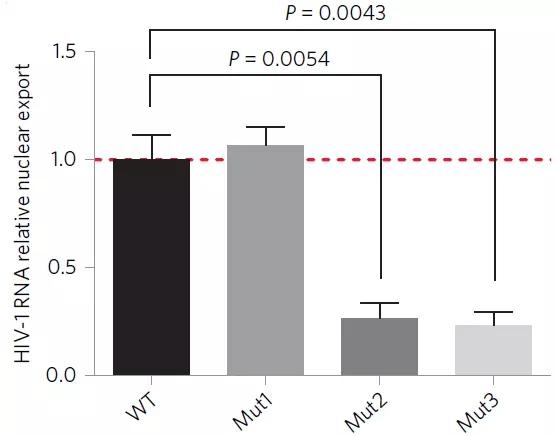
最后一个实验,作者对RRE上的A7877和A788进行突变,包括U7871C + A7877G (Mut1)、A7883G (Mut2)和两个位点同时突变(Mut3),然后侵染293细胞。A7877突变即Mut1,无论是NTC还是敲低METTL3、METTL14和ALKBH5都与WT无显著差别。无论是NTC还是三种甲基化酶的敲低,A7883突变(Mut2)或Mut3都会严重影响HIV-1的复制能力。A7877突变并不影响病毒出核效率,但是A7883突变改变了RNA二级结构以及与Rev互作的复合物结构,从而严重干扰病毒出核效率。所以A7883上的m6A修饰是RRE与Rev互作结合的关键所在,与病毒的复制及出核密切相关。
