- NCBI RefSeq
NCBI RefSeq的命名规则是以两个字母开头,后面跟“_”,然后是纯数字,常见的有“NM_”、“XM_”、“NR_”、“XR_”。有三点需要清楚:
1、“NM_”、“XM_”命名的记录代表的是编码基因,“NM_”对应“NP_”,“XM_”对应“XP_”;
2、“NR_”、“XR_”命名的记录代表的是非编码基因;
3、“XM_”,“XR_”通过计算机算法预测得到,而“NM_”和“NR_”都是有一定得实验数据支撑,但并不是说“XM_”和“XR_”就不存在于细胞中。NCBI RefSeq一直在更新,这些命名的记录代表的是一种状态,经常会碰到某个“XM_”记录被“NM_”代替,或者“NM_”记录由于缺少证据而从NCBI RefSeq删除。
更多命名含义如下图:

- Ensemble
- Ensembl Stable ID是来源于Ensembl数据库的编号系统。它的命名由三部分组成:[species prefix][feature type prefix][a unique eleven digit number]. (根据不同物种设置的前缀+数据所指类型【例如,蛋白质,基因】+一段特定的数字),
- 常见的物种前缀:ENS代表Homo sapiens (Human);ENSMUS代表Mus musculus (Mouse);ENSRNO代表Rattus norvegicus (Rat);
- 常见的数据类型:字母“G”代表gene,比如小鼠基因就命名为ENSMUSG###########;字母“T”代表transcript,比如ENSMUST###########;字母“P”代表protein,比如ENSMUSP###########。
- 有时有不同的版本, 则在 Ensembl ID 后面加上小数点和版本号(例如:ENSG00000223972.5)。如果要查询ENSG00000223972.4,则需要去其他release中查找,目前已经更新到release 97。
- pri-miRNA, pre-miRNA 和 mature miRNA的概念:成熟的miRNAs是由较长的初级转录物经过一系列核酸酶的剪切加工而产生的,初级转录物称为pri-miRNA。pri-miRNA长度从几百到几千个碱基不等,带有5‘帽子和3’polyA尾巴,以及1到数个发夹径环结构。Pri-miRNA经剪切产生约70个碱基的miRNA前体,即pre-miRNA。pre-miRNA经进一步剪切,形成长度约为22个碱基的单链成熟miRNA;
- 常见物种hsa,mmu和rno分别代表人,小鼠和大鼠;
- 在mirbase数据库中,pre-miRNA用mir表示,mature miRNA用miR表示;
- 绝大多数pre-miRNA可以产生两个mature miRNA,对应pre-miRNA茎环结构5‘和3‘序列的mature miRNA分别加后缀-5p和-3p以示区分,如rno-miR-325-5p和rno-miR-325-3p;
- 位于基因组不同部位但产生同样的mature miRNA的pre-miRNA在序号后添加短线和阿拉伯数字以示区别,如hsa-mir-199a-1, hsa-mir-199a-2;
- 高度同源的miRNA(microRNA)在数字后加上英文小写字母(a,b,c,)区分,如hsa-miR-34a,hsa-miR-34b,hsa-miR-34c等,通常他们的mature miRNA仅相差1-2个碱基,且他们的seed sequence相同,也就是说他们调控的靶基因相同;
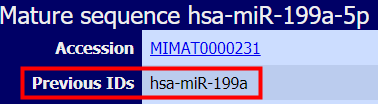
- 在一些miRNA与靶基因3UTR结合位点预测数据库有时会看到带有“*”的mature miRNA,比如hsa-miR-199a*,以前“*”表示对应的mature miRNA表达量低或者是次要产物,但是现在miRbase数据库已经取消这样的命名,即如果一个pre-miRNA有两个mature miRNA,用-5p和-3p以示区分。但是miRbase数据库会告知以前名称与现在名称的对应关系。

三个常用数据库的命名规则就到这里,了解数据命名规则特别是mirbase和Ensemble两个数据库可以很快速的知道基因信息对应的物种(比如人,小鼠,大鼠),数据类型(基因,转录本,蛋白),对于我们对信息准确与否的简单判断非常有帮助。

