教你怎么做miRNA靶基因验证(预测篇)
今天为大家分享一些miRNA研究的经验。分子生物学领域最高大上的研究莫过于发现分子间的直接相互作用机制了,不发现个分子间新的相互作用关系你都不好意思投稿高分文章。但是,相信大家都做过Co-IP,CHIP,EMSA甚至RIP实验,很多时候都会被各种阴性结果弄得有掐死老板的冲动吧。
miRNA大家都不陌生,作为一类生物体内的重要非编码小RNA,能够通过靶向调控许多基因从而在几乎所有方面都能发挥调控作用。miRNA研究在中国那是相当的火爆,近几年的只要是miRNA相关的SCI文章,作者几乎都是清一色的中国人。为什么精明的国人都像抢日本马桶盖一样抢着研究miRNA呢?因为发现miRNA和靶基因的直接相互作用关系相当容易有木有!

首先miRNA和靶基因的作用模式相对简单,有许多权威的数据库比如targetscan(http://www.targetscan.org/),miranda(http://www.microrna.org/microrna/home.do),miRDB(http://mirdb.org/miRDB/)等就可以进行预测,关键他们还是免费的(这点太重要了!)。
我们来看一下这些网站怎么使用:
首先是大名鼎鼎的targetscan: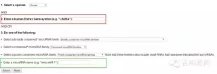
红框里填基因名字可以查询靶向的miRNA
绿框里填miRNA名字可以查询靶基因(注意要按照示例里的格式哦)
Miranda(又叫miRNA.org):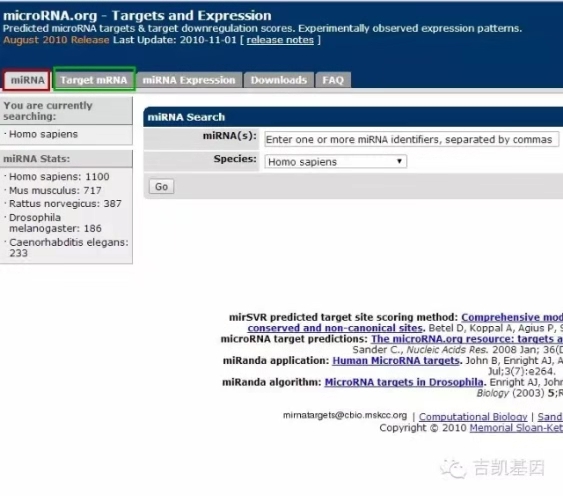
点红框可在跳出的页面填基因名字查询靶向的miRNA
点绿框可在跳出的页面填miRNA名字查询靶基因
还有miRDB: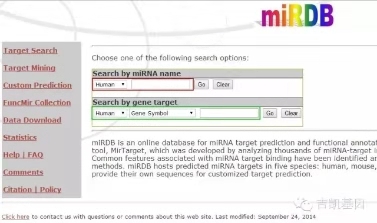
红框里填基因名字可以查询靶向的miRNA
绿框里填miRNA名字可以查询靶基因
不同数据库的计算方法不同,造成结果的差异,很多同学喜欢用好几个数据库的预测结果来个交集,哼哼,这么多算法都预测出来了害怕做不出?但是不要以为聪明人只有你一个哦,miRNA靶基因属于不可回收的珍惜资源,发表了就没了,经过10多年的研究,无数的论文已经基本上耗尽了这些优质的miRNA靶基因。现在大部分的文献都是只使用一个数据库,不是作者不愿用多种数据库的交集结果,而是很难再找到未被发表的优质新靶基因了。
通过使用免费的数据库预测出感兴趣的miRNA可能与哪些靶基因结合,随后就是用实验方法验证预测结果了。

