说到基因编辑,大家可能立马会想到crispr/cas9技术,原因不用多说,各种文章的发表已经让crispr/cas9技术“无所不能”。 基因编辑技术的数量种类和实现的可行性方面都取得了巨大的进步,基因编辑技术的出现和不断更新迭代使得研究复杂基因之间的相互作用成为可能,今天小编就如下基因编辑技术进行逐一介绍。
1、锌指核酸酶
2、TALENs
3、Cre-Loxp
4、CRISPR
5、Sleeping Beauty transposon
锌指核酸酶
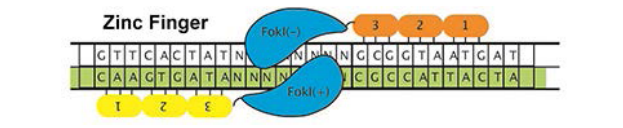
锌指核酸酶(ZFN)代表了迈向高效,有针对性的核酸酶的第一步。为了产生ZFN,设计了一系列锌指与特定基因组基因座结合,然后与FokI核酸酶融合。识别两个相邻位点的配对ZFN切割DNA,从而启动HDR。
最经典的锌指核酸酶是将一个非特异性的核酸内切酶FokI与含有锌指的结构域进行融合,其目的自然是对特定序列进行切割。锌指识别三联体,FokI核酸酶作为二聚体起作用,在两个不同ZF目标位点之间的间隔区切开DNA。被切开的DNA可以由切除的修复机制使切开处的单链部分被删除,然后又重新接到一起。
理论上讲,我们可以利用这种方法完成对染色体上特定片段的删除,从而达到构造突变体或完成治疗的工作。但是ZFN的效用受到合成时间长和非模块化组装工艺的限制。尽管计算工具有助于改善设计,但无法为每个基因组基因座设计合适的ZFN对。
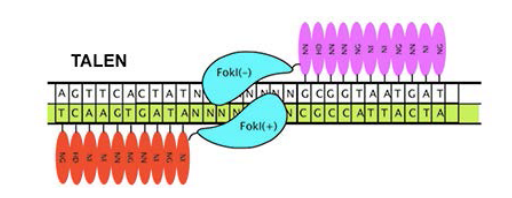
TALENs
TALENs在基因编辑技术上迈出了一大步。该模块系统基于从Xanthomonas spp分离的TAL效应子DNA结合蛋白与FokI核酸内切酶融合。经验丰富的科学家制作ZFN可能需要六个星期的时间,而TALENs技术的出现使得新手可以在短短几天内完成TALEN的构建。
TAL中心靶向结构域由33-35个氨基酸重复序列组成。这些重复由两个氨基酸组成,彼此不同,也就是它们的重复可变双残基(repeat-variable di-residue,RVD)。最终,正是这种RVD决定了TAL效应子(TALe)将识别出哪个单核苷酸:sHD靶向胞嘧啶,NI靶向腺嘌呤,NG靶向胸腺嘧啶和NN靶向鸟嘌呤。
由于ZF靶标仅限于由具有相应锌指的三联体组成的序列,因此平均每500bp基因组中有潜在的可靶向位点。TAL效应子有一些限制,例如目标必须以T开头,但它们仍然具有大约每35bp就可以找到潜在目标位点。
Cre-Loxp
Cre-loxp是一个非常有趣的工具,该工具可用于在转基因动物,胚胎干细胞和/或组织特异性细胞类型中创建特定的靶向DNA修饰。现已广泛用于控制基因表达,Cre重组酶在各种启动子的控制下或以其可诱导的形式对基因表达进行复杂的时空控制,尤其是在转基因小鼠研究中应用尤为广泛。
Cre-loxp系统由源自P1噬菌体的两个成分组成:Cre重组酶和loxP识别位点。
Cre重组酶最初命名是因为它“引起重组( causes recombination)”,后来也称之为“环化重组酶(cyclization recombinase)”),它是一个38 kDa的蛋白质,在loxP识别位点负责分子内和分子间的重组。该系统的主要优势在于Cre的作用不依赖任何其他辅助蛋白或辅因子,因此可广泛用于各种实验中。
LoxP((Locus of X(cross)-over in P1)则是位于P1噬菌体中的34bp序列,由两个13bp的反向回文序列和8bp的中间间隔序列共同组成,核心序列的不对称赋予了loxP位点具有方向性,典型的loxP序列为ATAACTTCGTATA-GCATACAT-TATACGAAGTTAT。loxP序列在除P1噬菌体之外的任何已知基因组中都不是天然存在的,并且由于足够长,几乎没有机会随机出现。因此,在DNA序列感兴趣的位置插入loxP位点可以进行非常特殊的操作。
具体原理:
Cre重组酶介导两个loxP位点之间的位点特异性重组事件,这些位点可以位于相同或不同的DNA片段上。单个loxP位点上的每个13
bp重复序列均被Cre蛋白识别并结合,形成二聚体。然后,两个loxP位点以平行方向排列,从而允许四个Cre蛋白形成四聚体。双链DNA断裂发生在每个loxP位点的核心间隔区内,然后两条链被连接,从而导致相互交换事件发生。根据loxP位点的位置和方向,以下三种情景:
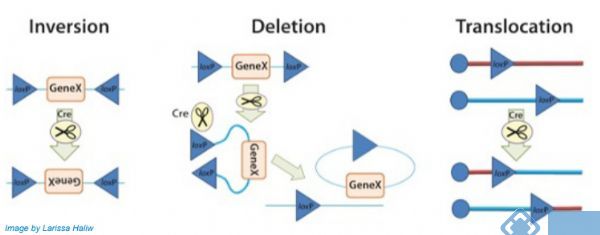
✫Inversion:当两个LoxP位点位于一条DNA链上但方向相反时,Cre重组酶能诱导两个LoxP位点间的序列翻转;
✫Deletion:当两个LoxP位点位于一条DNA链上且方向相同,Cre重组酶能有效地敲除两个LoxP位点间的序列;
✫Translocation:当两个LoxP位点分别位于两条不同的DNA链或染色体上,Cre重组酶能诱导两条DNA链发生交换或染色体易位;
However,从以上情景我们也可以看到在Cre酶存在时,三种情景的变化其实是可逆的,那么该如何使这种动态变化达到一种稳定状态呢?如下图所示,通过引入两对不同的LoxP位点,经过两组LoxP点的两轮重组我们即可达到一种稳定状态。也就可以通过Cre的存在与否来控制基因的表达了。
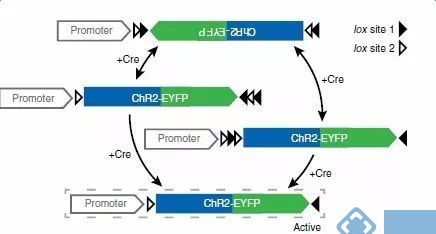
具体原理如下:
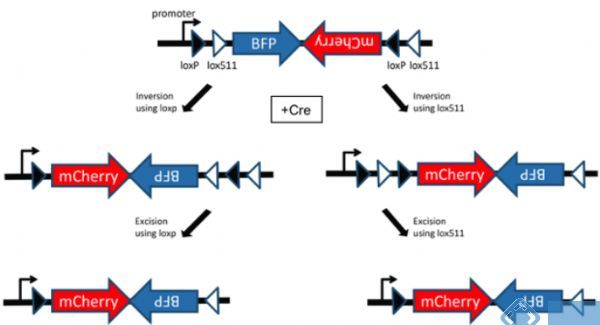
图中的白色箭头对应的是LoxP位点,黑色箭头对应的是Lox-2722位点。这两种位点都能被CRE所识别,并在相同位点间发生重组,而不同位点之间不能发生重组。
发生重组时,方向相反的两个位点之间的序列会被颠倒,而方向相同的两个位点之间的序列被切除。
在图中,假设第一次重组发生在LoxP位点之间,原本反向插入在启动子后的目的基因(不表达),重组后变成顺式结构,能够被启动子驱动表达。
第二次的重组发生在两个Lox-2722位点之间,它们的重组导致了中间的LoxP位点丢失,同时只保留下一个Lox-2722位点。
最后剩下的Lox-2722和LoxP位点之间不能发生重组,所以整个基因结构便被固定下来。
在这个基础上,也就产生了DO(Cre-off)、DIO(Cre-on)和Cre-Switch(Cre-off/on)。这三种系统中,LoxP序列在同一条载体上,并且方向相同, 只是外源基因的插入方向不同。
➩ DO系统(Cre-off)
插入基因与启动子方向一致,在Cre重组酶存在的情况下会发生重组导致基因方向反向,因此基因不表达,所以称之为Cre-Off;
➩ DIO系统(Cre-on)
插入基因方向与启动子方向相反,在Cre重组酶不存在的情况下不表达,只有当Cre酶存在时,发生重组使基因方向与启动子方向一致,才能使该基因表达,因此该系统称之为Cre-On;
➩ FLEX系统(Cre-on)
FLEX系统与DIO系统一致,只是LoxP位点方向与DIO系统不同。插入基因方向与启动子方向相反,在Cre重组酶不存在的情况下不表达,只有当Cre酶存在时,发生重组使基因方向与启动子方向一致,才能使该基因表达。
➩ Cre-Switch系统(Cre-off/on)
对于Cre-Switch来讲,则是在LoxP序列之间插入了两个阅读框,而这两个阅读框的方向相反,则可以通过Cre酶的存在与否来控制这两个基因的表达。
Cre-loxp系统说到这里,说它是一个非常有趣的工具,大家都认同的吧?Cre-LoxP系统是在神经系统中应用最广泛的条件性基因敲除工具,Cre-loxp系统优点如下:
◉ 高效性:Cre重组酶与具有LoxP位点的DNA片段形成复合物之后,可以提供足够的能力引发之后的DNA重组过程,重组过程简约高效;
◉ 特异性强:LoxP序列的唯一性,保证基因重组的特异性;
◉ 应用范围广:Cre重组酶可以在生物体不同的组织、不同的生理条件下发挥作用;
◉ 可由二型启动子启动表达:Cre重组酶的编码基因可由任何一种二型启动子驱动,由此保证Cre重组酶在生物体不同的细胞、组织、器官或者在不同的发育阶段或不同的生理条件下表达,从而实现较高的组织和细胞特异性。
Cre/loxp系统是一种完善的研究工具,正由于Cre/loxp系统的上述优点,尤其是在转基因小鼠领域。下面列出了一些最常见的用途:
◉ Cre依赖性基因表达:在目标基因上游放置两边都带有loxP位点的终止密码子(通常称为“ lox-stop-lox”或“ LSL”表达盒),在没有Cre重组酶的情况下,将阻止基因表达。在Cre存在的情况下,切除终止密码子,基因表达。
◉ Cre依赖性基因敲除:将loxP位点置于基因的两侧(称为“ floxing”,意为“flanked by loxP”),在Cre不存在时允许基因表达;当Cre存在时该基因表达将被破坏或删除。
◉ 选择标记的去除:在常规的小鼠编辑中,通常使用选择标记进行目标克隆筛选,但是在初始选择过之后,通常希望删除标记。此时通过对选择标记进行floxing处理,利用Cre可轻松执行此操作。
◉ 调节Cre表达:在发育的某些阶段通过将Cre置于特异性启动子下游,或通过将Cre置于诱导启动子(例如强力霉素诱导表达系统),这样Cre重组酶只能在指定的细胞中或指定的时间表达,同时结合上述一些loxP使用方法,可以进行不同要求基因修饰。
CRISPR
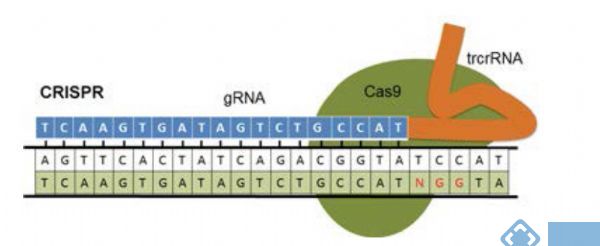
CRISPR是细菌免疫系统的重要组成部分,可使细菌记住并破坏噬菌体。在基因编辑应用中,Cas9核酸内切酶通过gRNA序列进行靶向,从而诱导双链断裂。像ZFN和TALENs一样,CRISPR/Cas9也使用HDR,但是使用RNA来指定编辑使得该系统更便宜,更省时,更精确。与TALENs相比,CRISPR的应用更为广泛,每周都会有使用该技术发表的新论文。
CRISPR/Cas9系统有两个不同的组成部分:(1)指导RNA(gRNA)和(2)核酸内切酶,在上图中为Cas9。当在细胞中表达时,gRNA/Cas9复合物通过gRNA之间的碱基配对募集到靶序列及其在基因组DNA中的互补序列。为了使Cas9成功结合到DNA,基因组DNA中的靶序列不仅必须与gRNA序列互补,而且还必须紧随其后的是正确的PAM(protospacer adjacent motif)序列。请注意,PAM序列存在于DNA靶序列中,但不存在于gRNA序列中,任何具有正确靶序列且后接PAM序列的DNA序列都将被Cas9结合。不同的cas9蛋白识别的PAM序列不同。
|
Cas9 Species |
PAM sequence (5’ to 3’) |
|
Streptococcus
pyogenes (Sp) |
NGG |
|
Staphylococcus
aureus (Sa) |
NGRRT or NGRRN |
|
Neisseria
meningitidis (Nm) |
NNNNGATT |
|
Streptococcus
thermophilus (St) |
NNAGAAW |
|
Treponema denticola
(Td) |
NAAAAC |
CRISPR/Cas9技术存在如下应用:
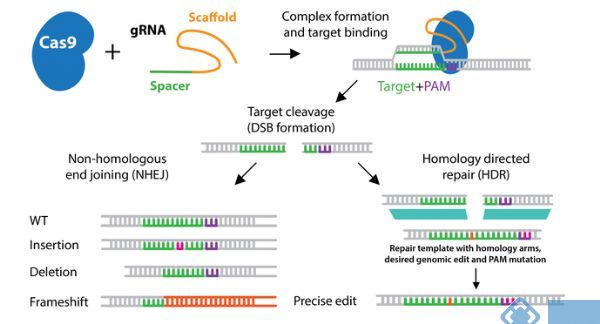
1、导致双链断裂:完整功能的CRISPR/Cas酶将根据gRNA序列在特定位置引入双链断裂(DSB)。DSBs优先通过非同源末端连接(NHEJ)在细胞中修复,该机制经常会导致DNA中引物插入或缺失(indels)。插入缺失会导致移码功能等位基因缺失。
2、导致单链断裂:CRISPR/Cas切口酶突变体导致Grna靶向的DNA单链断裂,而不是野生型Cas酶产生的双链断裂。使用切口酶突变体,需要两个gRNA,两个Grna位于DNA的两条链,且两个gRNA的位置距离合适。
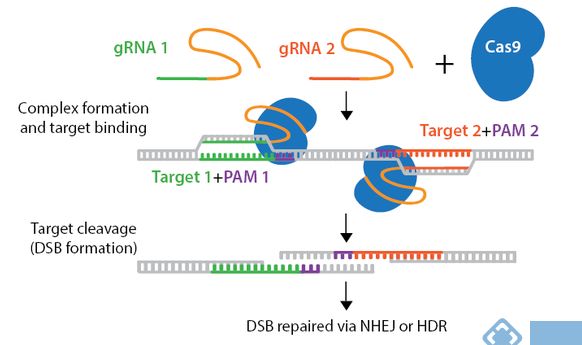
3、碱基编辑:将dCas9与胞苷脱氨酶蛋白融合,可以成为一种特定的碱基编辑器,可以改变DNA碱基而不会引起DNA断裂,该系统可以进行C-> T转换(或相反链上的G-> A转换)。
而2019年10月21日发表在Nature上面的文章"Search-and-replace genome editing without double-strand breaks
or donor DNA”,已经实现碱基任意转换与增删。
4. 激活上调基因表达:将dCas9与转录激活肽融合可以增加特定基因的转录。合理设计gRNA序列以将dCas9激活子引导至感兴趣的基因的启动子或调控区域,实现基因的上调。
5、干扰下调基因表达:将dCas9与转录抑制肽(如KRAB)融合,可以通过干扰转录来降低基因表达。
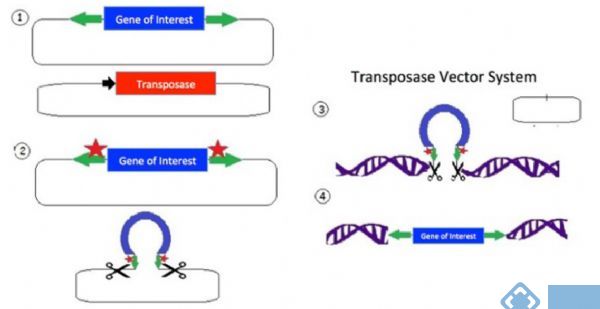
Sleeping Beauty transposon
睡美人转座子(Sleeping
Beauty transposon)系统由两个部分组成:1)SB转座酶,催化转座所需的酶;
和2)含有可在基因组内移位的基因表达盒的转座子。睡美人转座子被证明是无需使用病毒载体即可产生特定基因突变和基因破坏的令人兴奋的工具。睡美人转座子同样可用于基因组编辑,可以作为CRISPR/Cas9技术的一种有价值的替代工具,并且睡美人转座子的使用可能会在未来几十年内有增长。
如上图,(1)转座子由位于质粒主链中目标基因(蓝色)侧翼的一组反向重复序列(绿色)组成。第二个质粒包含用于表达转座酶的转座酶基因(红色)。
(2)转座酶表达(红色星号)并结合反向重复序列(绿色);发生核酸内切酶反应,切割DNA。
(3)释放的转座子可以与带有TA二核苷酸DNA链结合(在人类基因组中有许多这样的位点)。除去转座子后,原始质粒为空,然后质粒被细胞降解。
(4)转座酶在DNA中产生双链断裂,并允许转座子整合。
借助随手可得的多种强大有效的基因编辑方法,我们已经进入了基因编辑的黄金时代。相信在以后会有越来越多的工作将注重于完善这些技术,以确保高特异性和高活性。
参考资料:
1. Genesis. 2000
Feb;26(2):99-109. Cre recombinase: the universal reagent for genome
tailoring
2. Nature. 2019 Oct 21.
doi: 10.1038/s41586-019-1711-4. Search-and-replace genome editing without
double-strand breaks or donor DNA
3. Plasmids 101: A
Desktop Resource Created and Compiled by Addgene March 2017 (3rd Edition)
4. CRISPR 101: A
Desktop Resource Created and Compiled by Addgene May 2017 (2nd Edition)