小RNA测序(miRNA-seq)
产品名称: 小RNA测序(miRNA-seq)
英文名称: miRNA-seq
产品编号: yb004
产品价格: 5000
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
- 联系人 : 谷小姐
- 地址 : 上海市闵行区浦江镇联航路1239弄8B号3楼
- 邮编 : 201112
- 所在区域 : 上海
- 电话 : 181****9791 点击查看
- 传真 : 点击查看
- 邮箱 : gulingzhi@yingbio.com
miRNA测序
小RNA是一类重要的体内调节分子,主要包括miRNA、piRNA和siRNA。它的功能主要是诱导基因沉默,参与基因转录后调控,从而调节细胞生长、分化,以及个体发育、生殖等重要生物学过程。小RNA测序技术采用胶分离技术,收集样品中18-30nt的RNA片段,利用高通量测序技术,一次性获得单碱基分辨率的数百万条小RNA序列信息,依托强大的生物信息分析平台,鉴定已知小RNA,并预测新的小RNA及其靶标基因。
技术优势
· 通量高:一次测序得到500万条以上的序列;
· 分辨率高:可以检测小 RNA单个碱基的差异;
· 精准度高:从几个到数十万个拷贝精确计数;
· 不依赖已知信息:既能鉴定已知小RNA,又能发现新小RNA;
· 可重复性高:深度测序保证了抽样随机性,重复性非常好,无需重复实验。
研究内容
基于Illumina HiSeqTM 2000高通量测序技术的小RNA数字化分析,采用边合成边测序的方法,可减少二级结构造成的区域缺失。该技术可以对高质量序列进行序列长度分布的统计及样品间公共序列统计,将筛选后的高质量序列分类注释,从而获得样品中包含的各组分及表达量信息,并对所有小RNA片段进行注释,对新的miRNA则进行靶基因预测。
一、标准信息分析
A. 数据产出统计,对原始测序数据去接头污染,去低质量reads;
B. 18~30 nt 小RNA 测序结果的长度分布;
C. 样品间的公共序列和特异序列的分析;
D. 小RNA在选定的参考基因组上的分布;
E. 小RNA与rRNA、tRNA、snRNA、snoRNA的比对信息;
F. 小RNA与重复序列的比对信息;
G. 小RNA与外显子或内含子的比对信息;
H. 小RNA与miRBase 中指定范围的已知的 miRNA的比对;
I. 按照优先级将小RNA进行分类注释;
J. 利用 Mireap 对没有注释的small RNA进行预测,预测新的miRNA,绘制新的miRNA 的二级结构图;
K. 已知miRNA的表达谱构建;
L. 已知miRNA 的家族分析。
二、高级信息分析(基于标准分析)
A. 已知miRNA和novel miRNA的靶基因预测;
B. 已知miRNA和Novel miRNA靶基因GO注释和KEGG;
C. 已知miRNA的碱基编辑分析;
D. 已知 miRNA 差异分析和聚类分析;
E. Novel miRNA 差异分析(2个或2个以上样品)和聚类分析;
F. 差异miRNA 靶基因预测;
G. 差异miRNA 靶基因GO 注释和KEGG 通路分析。
案例:通过深度测序分析乳腺癌中microRNA的序列和表达
背景: microRNAs调控很多对肿瘤发生非常重要的基因。乳腺癌中很多microRNAs是下调的,但系统的乳腺癌中microRNA的表达分析还没有被报道。
目的: 利用Illumina HiSeq2000测序平台对不同类型乳腺癌组织和细胞进行测序,分析其microRNAs表达差异。
结果:通过对11个正常乳腺组织,17个非转移组织,151个转移组织,及6种乳腺癌相关细胞系测序分析发现,正常组织中miR-21是高表达的,发生转移的乳腺癌病人的乳腺组织中mir-423是上调的,三阴乳腺癌病人中mir~17-92是特异性上调的,但是这些变化并不显著。这些结果表明,基于microRNAs水平对乳腺癌类型进行区分及预测转移统计学上可能是可行的,但是乳腺癌类型及转移的差异并不是由于富集的microRNAs的下调驱动的,提示在乳腺癌的发展过程中起作用的microRNAs不多。
原文索引:
Farazi TA1, Horlings HM, Ten Hoeve JJ(2011)MicroRNA sequence and expression analysis in breast tumors by deep sequencing. Cancer research, 71(13), 4443-4453
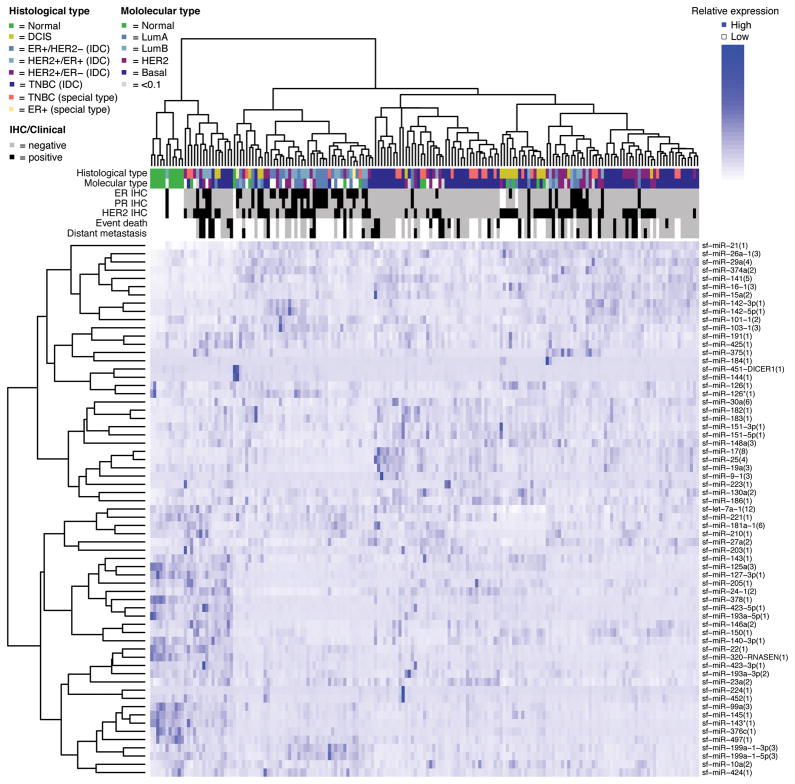
图1: 病人样品的microRNAs的聚类分析

图2:microRNAs与mRNAs的聚类分析的比较
