环境微生物群落多样性分析,基于Roche 454 FLX +、Illumina MiSeq等第二代高通量测序平台,对核糖体RNA高变区域,比如16S/18S/ITS等序列;或功能基因,比如细菌和古菌的氨氧化酶基因进行测序,揭示环境样品中众多不同类型微生物种类以及它们之间的相对丰度和进化关系。探讨微生物多样性,对于研究微生物与环境的关系、环境治理和微生物资源利用有着重要的理论和现实意义。
美吉优势
★ 采用定向测序技术,避免了非定向测序造成的数据冗余,能充分利用每条有效序列,且便于后期的菌群分类处理。
★ 开发了一套稳定的多样本平行测序实验方案,并解决了样本间数据量不平衡的技术难题,一次可实现200个样品的平行测序。
★ 环境微生物群落多样性分析有Roche 454 FLX +和Illumina MiSeq两种高通量测序平台可供选择,根据不同的测序要求提供不同的解决方案。
★ 众多的成功案例,使美吉生物在环境微生物群落多样性测序及分析方面处于国内**地位。
技术路线

生物信息分析流程
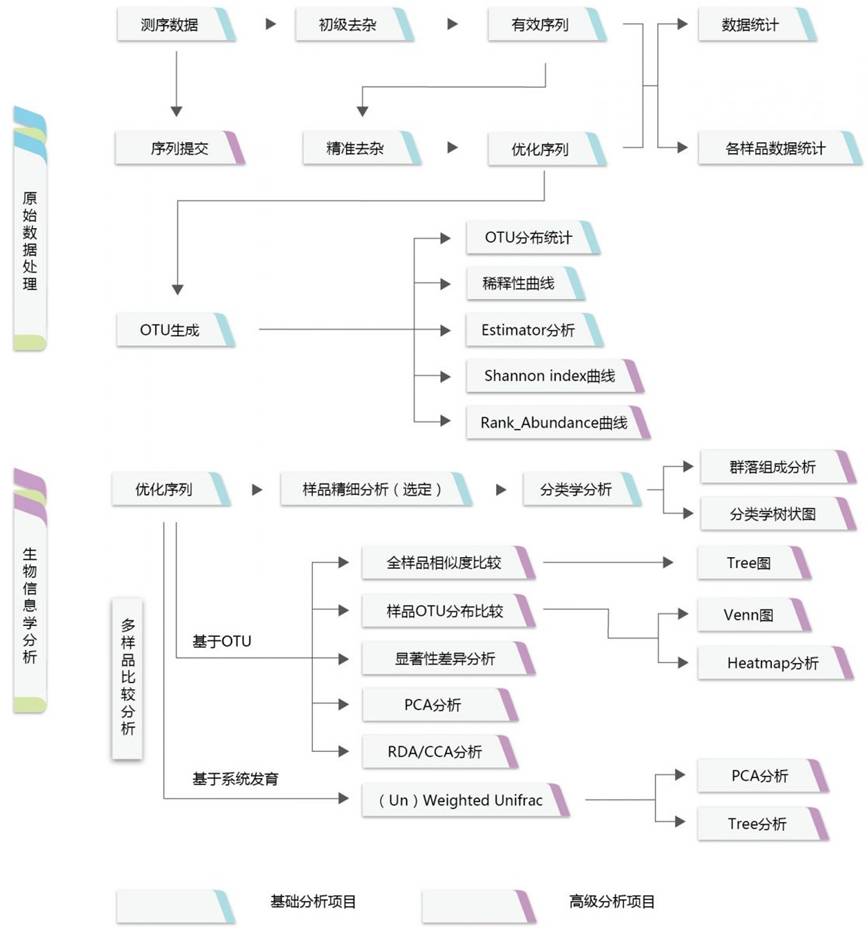
研究内容
1. 数据统计分析
2. 数据根据Barcode信息回归样品
3. OTU(Operational Taxonomic Units)聚类
4. 稀释性曲线(Rarefaction Curve)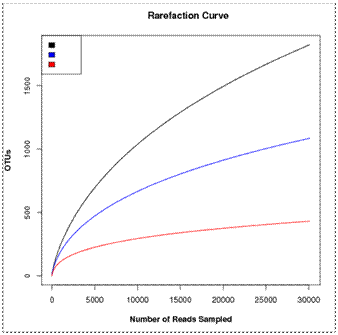
稀释性曲线
5. 分类学分析(Silva数据库)
6. 菌群多样性、丰度和测序深度指数计算
7. 多样品间两两sharedchao、sharedace指数计算
8. 组间显著性差异分析
9. 群落结构分析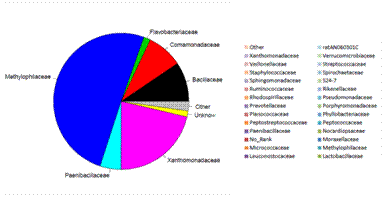
单样品群落结构饼图
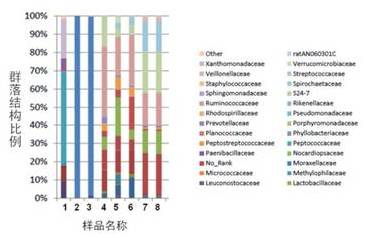
多样品群落结构柱状图
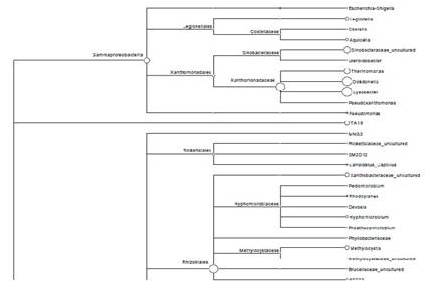
10. 分类学树状图