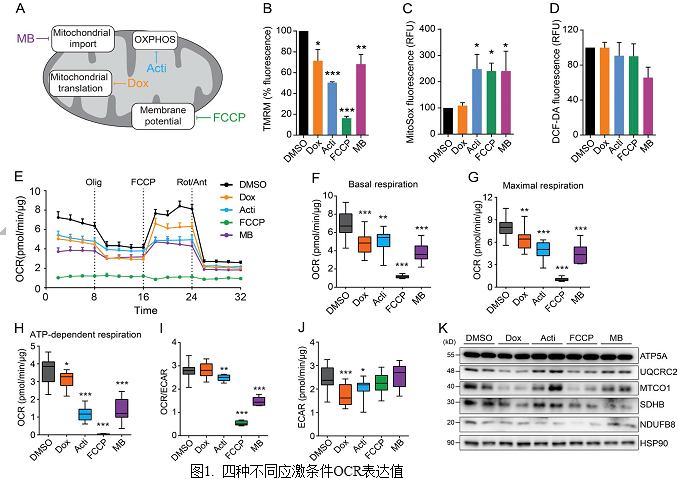
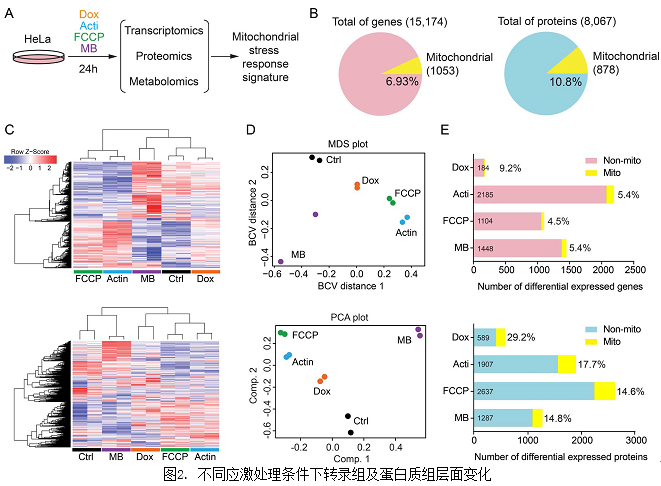
2. 不同线粒体应激处理后转录组、蛋白质组、代谢组变化
作者分别用四种物质(doxycycline四环霉素、actinonin放线酰胺素, FCCP碳酰*-4-三氟甲氧基苯腙,和MB)对Hela细胞进行处理,时间24h,后通过转录组测序,蛋白质质谱分析(TMT)及高通量非靶向代谢组检测技术(Figure 2A)。从转录组及蛋白质的分析结果来看,共鉴定出15174 转录本及8067个蛋白质,其中线粒体中的差异基因及蛋白分别占到1053及878个,占比分别为6.9%及10.8%。通过聚类分析结果显示,与actinonin放线酰胺素, FCCP碳酰*-4-三氟甲氧基苯腙及MB诱导结果相比, doxycycline诱导效应相对较弱,不是很显著(Figure 2C 和D)。
在FDR<0.05的参数设置下,对不同诱导条件下的差异表达基因及蛋白进行统计分析鉴定(Figure 2E)。结果显示,actinonin放线酰胺素和MB诱导条件下,转录组水平的诱导响应较为显著,而在doxycycline四环霉素及FCCP碳酰*-4-三氟甲氧基苯腙的诱导下的Hela细胞,其蛋白质组水平诱导较为显著。同时作者发现,在所有的诱导条件下,线粒体水平上的差异变化,蛋白质组层面比转录组层面的变化较为显著,其线粒体差异基因表达量百分比约为其他基因的3倍左右(Figure 2E)。有效的证明了不同应激处理条件下线粒体在转录组层面及蛋白质组层面发生了有效的变化。
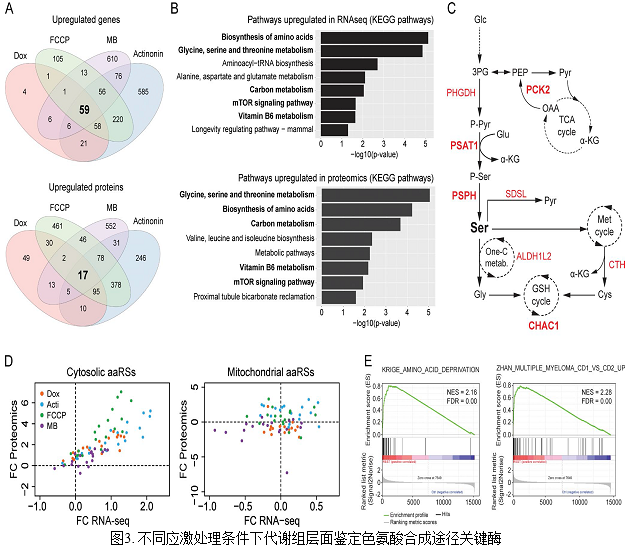
3. 不同线粒体应激处理导致氨基酸代谢途径及生物合成途径发生变化
作者通过对上调的基因和蛋白质进行分析验证,发现59个基因上调,只有17个蛋白质上调(Figure.3A ),上调主要发生在转录组水平,并且集中在线粒体基因的上上调数为4个genes,2个蛋白质,说明了压力诱导产生的表达量变化不集中在线粒体层面上。作者通过转录组及蛋白质组的KEGG富集分析结果中发现,主要集中与氨基酸代谢途径(Fig.3B),尤其是丝氨酸代谢途径相关的生物合成过程,并发现色氨酸合成过程上调的关键酶主要是PSAT1、PSPH及PHGDH以及SDSL。
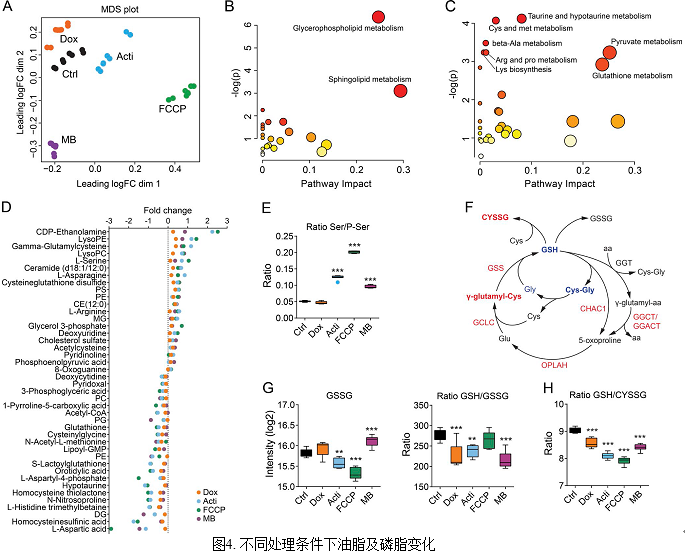
4.不同线粒体应激处理改变细胞代谢产物
作者通过代谢组方法,研究4种不同处理条件下细胞代谢产物的变化。代谢组数据鉴定到约300个有注释的代谢物质,通过聚类分析及热图的分析结果显示,代谢产物趋势与转录组和蛋白质组整体上趋势保持一致。其中doxycycline四环霉素处理组产生的影响效果更加显著。通过差异表达分析及富集分析显示:不同应激处理后响应效果最明显的为甘油磷脂及鞘磷脂代谢途径(Figure.4B),并发现四种应激处理后明显上调或下调的代谢产物基本保持一致,包括ceramide神经酰胺,CDP-ethanolamine乙醇胺,及几种溶血磷脂酰胆碱lysophosphatidycholine 、lysophosphatidylethanolamine(Fig. 4D)。这些代谢产物的变化充分说明了丝氨酸代谢增加通过神经酰胺的效应有效促进了油脂及磷脂质的生物合成。
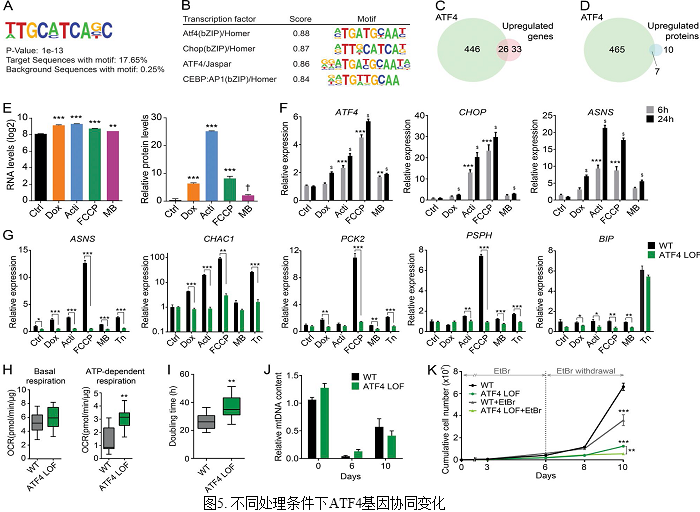
5. ATF4基因与不同应激处理协同变化
为了推测应激处理响应变化关键因素,作者采用HOMER分析方法对共同存在的上调基因进行Denovo motif分析,并得到高分motif,序列为5′-TTG CAT GACG-3′,其在靶基因中存在比例为17.65%,与ATF4、DDIT3/CHOP及CEBPB序列相似,其中ATF4得分率最高,极有可能是对应的转录因子(Figure.5B)并经免疫共沉淀方法进行验证,同时进行验证的还有CHOP。
作者同时发现约一半的由应激处理响应的基因同样是ATF4转录因子对应的靶基因(Figure.5C)。为进一步验证,作者采用CRISP-Cas9敲除技术,获得ATF4缺失Hela细胞后,对其进行应激处理,发现其对应ASNA,CHAC1,PCK2,PSHP靶基因对应的缺失,并增强了ATP呼吸依赖性,说明其在线粒体功能调控上起重要作用。
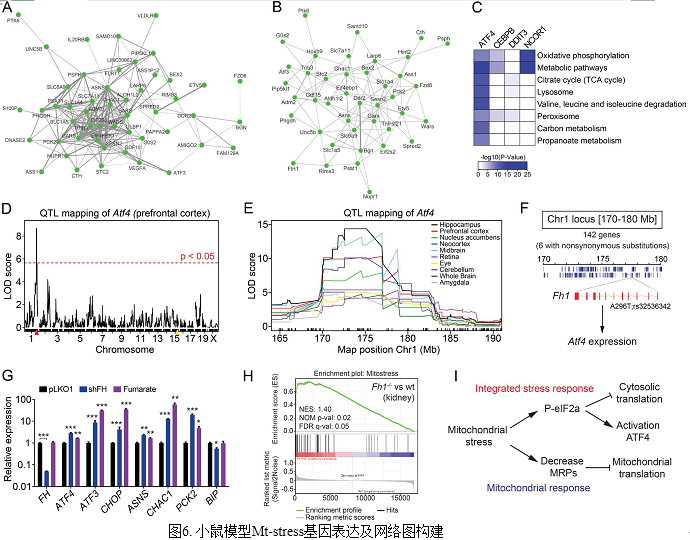
6. 比较分析人及模型鼠群中ATF4途径及线粒体胁迫间的遗传效应
作者通过GTEx 小鼠居群数据进行mt-stress基因表达研究,并构建网络分析图,并对ATF4,DDIT3/CHOP和CEBPB转录因子做关联分析,结果显示mt-stress基因与BXD数据聚类关系较近,通过系列的验证最终证明ATF4通过ISR激活效应调节细胞内代谢及线粒体功能,进而对外界胁迫刺激进行响应适应。
文章小结
通过转录组,蛋白质组及代谢组技术的单独测序及关联分析,对Hela细胞在四种试剂诱导下的样品进行线粒体功能胁迫方面的分析比较,初步探讨了线粒体应激胁迫响应途径参与的相关基因,并发现ATF4是响应的关键因子,并通过在人及动物鼠模型中进一步的验证比较寻求其调控机制,ATF4通过降低MRPs的表达水平,从而达到减弱转录能力的效果,从而有效适应外界胁迫条件,为研究线粒体功能障碍等相关疾病的研究探索提供了理论依据。
解析文献
Quirós P M, Prado M A, Zamboni N, et al. Multi-omics analysis identifies ATF4 as a key regulator of the mitochondrial stress response in mammals[J]. Journal of Cell Biology, 2017, 216(7):2027.
参考文献
1. Anders, S., P.T. Pyl, and W. Huber. 2015. HTSeq: A Python framework to work with high-throughput sequencing data. Bioinformatics. 31:166–169. http://dx .doi .org /10 .1093 /bioinformatics /btu638
2. Baker, B.M., A.M. Nargund, et al. 2012. Protective coupling of mitochondrial function and protein synthesis via the eIF2α kinase GCN-2. PLoS Genet. 8:e1002760. http ://dx .doi .org /10 .1371 /journal .pgen.1002760
3. Calvo, S.E., K.R. Clauser, and V.K. Mootha. 2016. MitoCarta2.0: An updated inventory of mammalian mitochondrial proteins. Nucleic Acids Res. 44(D1):D1251–D1257. http ://dx .doi .org /10 .1093 /nar /gkv1003
4. Kim, H.E., A.R. Grant, M.S. Simic, R.A. Kohnz, D.K. Nomura, J. Durieux,C.E. Riera, M. Sanchez, E. Kapernick, S. Wolff, and A. Dillin. 2016. Lipid biosynthesis coordinates a mitochondrial-to-cytosolic stress response. Cell. 166:1539–1552.e16. http ://dx .doi .org /10 .1016 /j .cell .2016.08 .027
5. Mariño, G., F. Pietrocola, et al. 2014. Regulation of autophagy by cytosolic acetyl-coenzyme A. Mol. Cell. 53:710–725. http ://dx .doi .org /10 .1016 /j .molcel .2014 .01 .016