
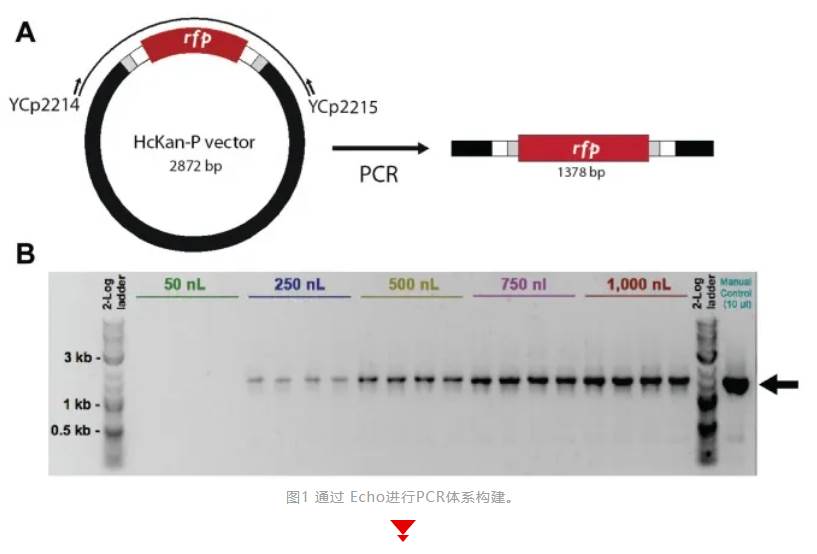
声学微滴喷射(AED)技术使用聚焦的声能实现高精度和准确度转移纳升级液滴。Echo这种非接触式、无需吸头、低体积加液技术将交叉污染的可能性降至最低,同时极大程度降低试剂和耗材的成本。迄今为止,Echo除了广泛应用于高通量化合物筛选之外,还有另一个强大的技术应用方向——快速发展的合成生物学领域,如DNA合成和连接,Echo声波移液使得PCR以及Golden Gate 和 Gibson两种一步法DNA连接方法成功地缩小至纳升级规模,极大地提高DNA合成和连接效率并有效将试剂成本降低20至100倍。大部分DNA组装的费用成本是酶,包括DNA聚合酶,因此,将反应体积从微升缩小到纳升级,同时保持高装配效率,将使合成生物学家更容易完成DNA合成和组装。这也使Echo声波移液成为DNA生物铸造厂时代的合成生物学中的一项工具性技术。比如在常规终点法PCR实验中,Echo可将PCR反应体系降低至250nL,成功扩增出1.3kb的片段。
Gibson DNA连接方法是合成生物学中使用最多的技术,它可以组装从DNA序列至小基因组重叠DNA片段大小,优点是它与序列无关并生成无痕DNA连接产物。Golden Gate DNA连接方法利用TypeIIS限制性内切酶和连接酶组合来组装DNA片段。正如我们先前所介绍的一样,使用Echo声波移液,能够实现这两种一步式DNA连接反应的降本增效,可在250 nL、500nL和1000 nL反应体积下实现Gibson的正确组装,效率可与手工实验相媲美或优于20 μL反应,这使试剂成本降低20倍以上;Golden Gate可实现50 nL 、250 nL和500 nL规模反应体积的DNA(手工实验时通常为 15 μL反应)成功组装,效率同样高于手工实验对照,使试剂用量至少降低30倍。
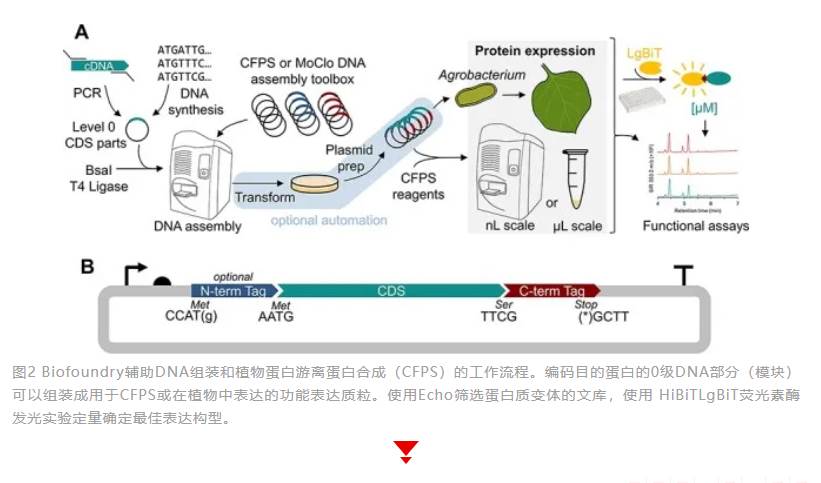
生物合成途径的阐明和重构以及调控回路和网络的工程设计,都需要蛋白质功能的知识。植物一直被用作生物活性和高价值天然产物的来源,但由于植物之中大型基因家族的普遍存在,使得将特定功能与单个蛋白质联系起来具有挑战性,同时也面临需要付出大量努力来优化表达和纯化方案进行蛋白质表征这一技术瓶颈。Biofoundry的独特优势能够加速“设计-构建-测试-学习”周期的能力,可加速优化并实现酶活性的快速筛选;无细胞蛋白质合成(CFPS)结合了DNA模板、能量源、氨基酸、核苷酸三磷酸 (NTP) 和过量辅因子,以及含有翻译机制的粗裂解物,是一种成熟的体外快速蛋白质生产工具,显著优势是能够直接进行功能分析,而无需耗时的细胞破碎和纯化方案,同时还适用于自动化和小型化,减少操作错误和CFPS反应中的可变性,促进主动学习引导的反应条件优化,并普遍增加实验的通量,最大限度地减少DBTL循环中的“构建”时间。SynTrack是一个基于web的工作流程驱动的bioCAM平台,用于管理和跟踪DNA制造过程。SynTrack导入boost指定的构建过程信息,其中包括操作人员(利用机器人平台)执行所有“构建”操作的分步说明。SynTrack可以为液体处理机器人(如Biomek FX和Echo平台)生成移液指令,自动将接收到的DNA片段重新排列到孔板中。通过Echo自动化平台构建2μL反应体积(手动为20μL)进行一步法Golden Gate DNA组装反应,随后构建2μL CFPS反应体系进行蛋白表达,通过荧光素酶(该标签可轻松去除)快速定量蛋白表达水平,随后无需蛋白纯化即可使用游离蛋白质合成反应产物进行多种功能表征。
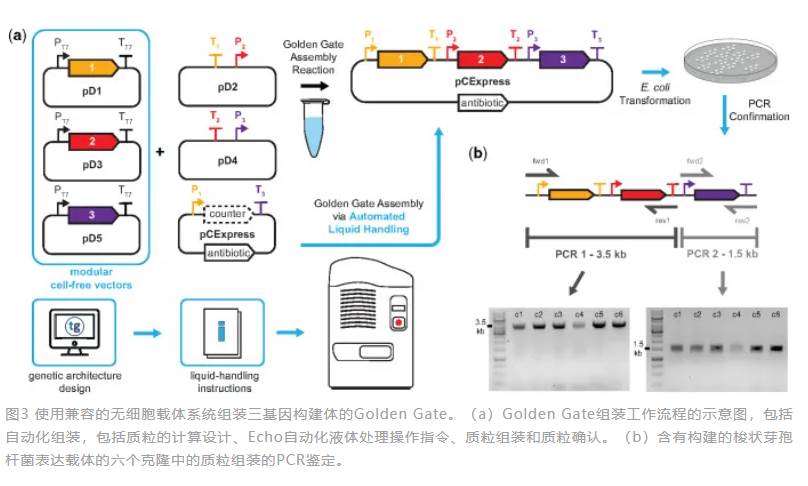
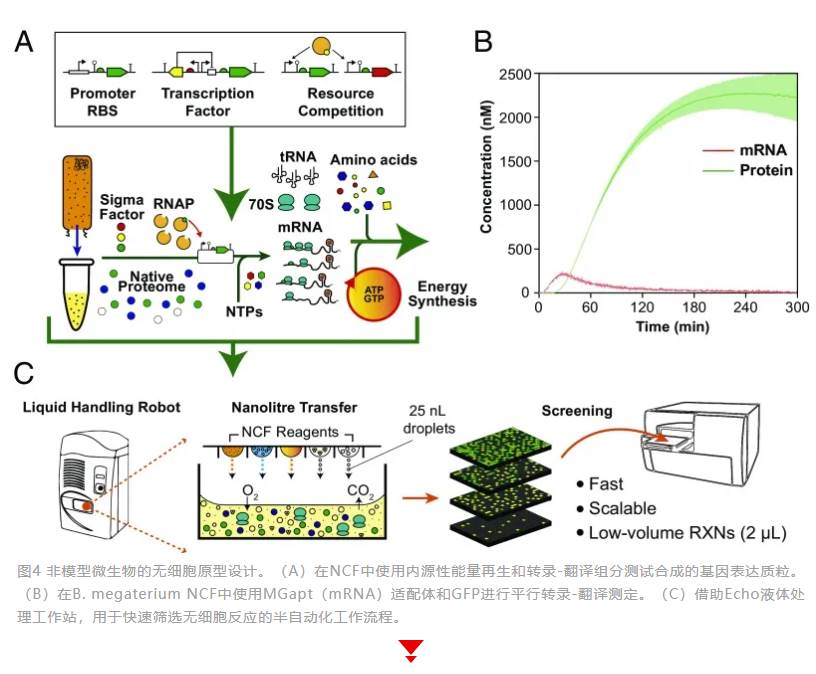
使用Echo声波移液配合无细胞蛋白合成(CFPS)技术也可在96和384孔板中进行代谢工程检测,通过Wood-Ljungdahl通路耦合的反向β氧化(r-BOX)通路,实现如C4-C6酸和醇生化产物的负碳合成,具有降低全球CO2排放的巨大应用潜能。正是由于模式生物大肠杆菌无细胞转录-翻译(TXTL)在DNA定向体外蛋白质合成的应用范围越来越大,一些实验室也开发了全大肠杆菌TXTL工具箱,并通过Echo实现了流程自动化。另一个无细胞合成生物应用方向是非模式生物体外原型设计和快速表征代谢途径,加速体内生物合成途径测试,使例如梭状芽胞杆菌等非模式生物能够利用可再生资源(木质纤维素生物质或CO和H2合成气)生产更多高价值产品。使用DNA组装设计自动化软件J5进行结构设计和生成实验运行worklist,发送至Echo自动化平台以2 μL体积进行Golden Gate DNA组装,可同时进行多达6个部分(三个独特启动子和终止子序列的开放阅读框)的组装,效率高达90%;这一质粒系统将为非模式生物提供体外和体内生物合成途径的简单测试框架,从而缩短开发周期。非模式生物巨双歧杆菌的天然无细胞(NCF)转录翻译平台的快速建立也借助了Echo,并和贝叶斯模型参数推断方法相结合,可以在数小时内对基因表达工具进行严格的表征,并且这个平台还有望扩展到一系列其他重要的底盘微生物的合成生物学应用。
微生物系统中异源生产的特定高价值化学物质大多需要资源密集型分析过程(如HPLC和MS)进行定量,较低的分析通量使DBTL循环遇到了一定的瓶颈。而生物传感器通常与基因表达系统耦合,为此人们越来越关注开发荧光生物传感器用于合成生物学中,来检测小分子和物理信号。通过为Echo编写Python移液脚本,构建384或1536孔板的10μL或2μL荧光酶标检测体系,基于大肠杆菌双键还原酶(EcCurA)非特异性结合后荧光极大增强的特性,开发了一种荧光复合物直接蛋白质(DiPro)生物传感器,作为微生物异源姜黄素酶促合成的检测单元,且不需要任何额外的基因表达后步骤或特定的蛋白质成熟要求。同样,使用Echo进行小反应体系的荧光素酶化学发光体系构建,高通量筛选分析脯氨酰寡肽酶异源表达变异株,将金属响应开关整合到蛋白质中,也为精确调节蛋白质功能提供了新的机会。
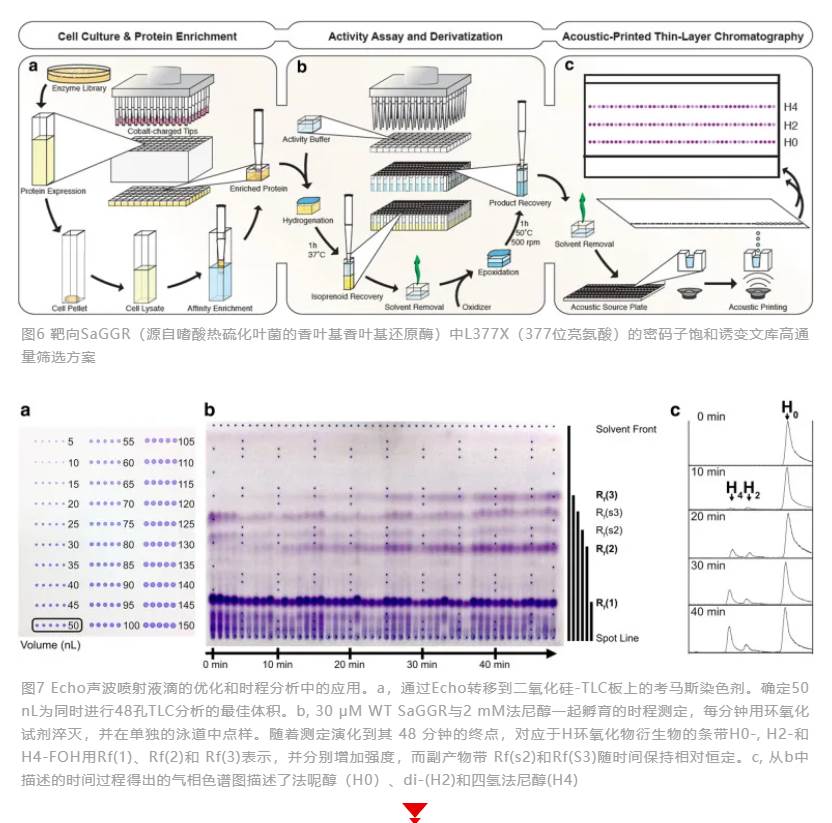
随着合成生物学的发展,使得通过大肠杆菌或酿酒酵母等微生物用于筛选氢化烯烃的酶,成为克服石油精炼过程中烯烃加氢面临的催化剂中毒、传质限制、发热以及与氢气、储存和催化剂等相关的一个重要的解决方案。相比于LC/GC-MS复杂的工作流程和较长的分析时间,使用Echo声波移液和薄层色谱搭建的自动化96孔筛选平台极大地提高了检测速度,使并行多个样品检测即时可视化,读值结果更可靠,成为一种高效的筛选流程,用于确定与工程化GGR蛋白文库相关的酶活性和产物形成谱。使用Echo将环氧化反应产物“打印”涂覆在薄层二氧化硅上,根据环氧化程度进行色谱分离,并与发色团共价连接,从而可以检测具有独特产物分布或增强还原酶活性的酶变体。经过GC-MS验证,这种基于薄层色谱的筛选可以区分选定突变体酶活性的四倍差异。
Echo高通量DNA组装还被用于在酿酒酵母固氮基因组合文库中筛选功能性固氮酶异源表达基因,为固氮谷物开发并改变全球农业系统提供极大支撑。DNA组装和Echo自动化液体处理结合技术也入选了高校合成基因组暑期课程,这一培训课程旨在向研究人员传授合成生物学和合成基因组科学最新进展背后的理论和实践技能,通过软件研讨会、课程和该领域领先成员的研究讲座,与会者能够在了解相关原理和技术的同时,在基于实验室的实践课程中直接使用合成生物学平台开发的软件和载体进行验证。在课程具体实践中,Echo自动化DNA组装技术大大提高了用于设计β-胡萝卜素异源通路文库的表达检测的通量。同时,使用该平台进行的番茄红素代谢途径的自动化设计和构建成果也于2020年发表。

通过以上案例我们可以发现,Echo声波移液在DBTL循环中起到承上启下的串联角色,既可接受“设计”流程中基于集成运算生成的脚本指令完成DNA组装,撑起“构建”阶段一众重要的应用方向;又可在“测试”过程中实现高通量自动化以及小体系精准检测,并传递数据为“学习”阶段提供支撑,因而是合成生物学和Biofoundry不可或缺的好帮手。
● 参考文献:
1、Kanigowska P, Shen Y, Zheng Y, Rosser S, Cai Y. Smart DNA Fabrication Using Sound Waves: Applying Acoustic Dispensing Technologies to Synthetic Biology. J Lab Autom. 2016 Feb;21(1):49-56. doi: 10.1177/2211068215593754. Epub 2015 Jul 10. PMID: 26163567; PMCID: PMC4814025.
2、Dudley QM, Cai YM, Kallam K, Debreyne H, Carrasco Lopez JA, Patron NJ. Biofoundry-assisted expression and characterization of plant proteins. Synth Biol (Oxf). 2021 Sep 11;6(1):ysab029. doi: 10.1093/synbio/ysab029. PMID: 34693026; PMCID: PMC8529701.
3、Cai YM, Carrasco Lopez JA, Patron NJ. Phytobricks: Manual and Automated Assembly of Constructs for Engineering Plants. Methods Mol Biol. 2020;2205:179-199. doi: 10.1007/978-1-0716-0908-8_11. PMID: 32809200.
4、Vögeli B, Schulz L, Garg S, Tarasava K, Clomburg JM, Lee SH, Gonnot A, Moully EH, Kimmel BR, Tran L, Zeleznik H, Brown SD, Simpson SD, Mrksich M, Karim AS, Gonzalez R, Köpke M, Jewett MC. Cell-free prototyping enables implementation of optimized reverse β-oxidation pathways in heterotrophic and autotrophic bacteria. Nat Commun. 2022 Jun 1;13(1):3058. doi: 10.1038/s41467-022-30571-6. PMID: 35650184; PMCID: PMC9160091.
5、Garenne D, Thompson S, Brisson A, Khakimzhan A, Noireaux V. The all-E. coliTXTL toolbox 3.0: new capabilities of a cell-free synthetic biology platform. Synth Biol (Oxf). 2021 Aug 4;6(1):ysab017. doi: 10.1093/synbio/ysab017. PMID: 34712841; PMCID: PMC8546610.
6、Karim AS, Liew FE, Garg S, Vögeli B, Rasor BJ, Gonnot A, Pavan M, Juminaga A, Simpson SD, Köpke M, Jewett MC. Modular cell-free expression plasmids to accelerate biological design in cells. Synth Biol (Oxf). 2020 Oct 14;5(1):ysaa019. doi: 10.1093/synbio/ysaa019. PMID: 33344777; PMCID: PMC7737004.
7、Moore SJ, MacDonald JT, Wienecke S, Ishwarbhai A, Tsipa A, Aw R, Kylilis N, Bell DJ, McClymont DW, Jensen K, Polizzi KM, Biedendieck R, Freemont PS. Rapid acquisition and model-based analysis of cell-free transcription-translation reactions from nonmodel bacteria. Proc Natl Acad Sci U S A. 2018 May 8;115(19):E4340-E4349. doi: 10.1073/pnas.1715806115. Epub 2018 Apr 17. PMID: 29666238; PMCID: PMC5948957.
8、Kennedy A, Griffin G, Freemont PS, Polizzi KM, Moore SJ. A curcumin direct protein biosensor for cell-free prototyping. Eng Biol. 2022 Aug 18;6(2-3):62-68. doi: 10.1049/enb2.12024. PMID: 36969103; PMCID: PMC9996706.
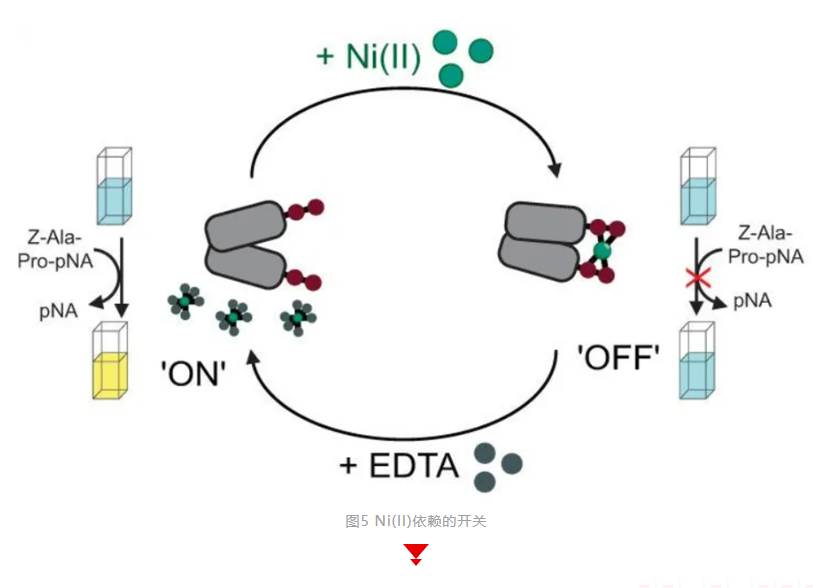
9、Zubi YS, Seki K, Li Y, Hunt AC, Liu B, Roux B, Jewett MC, Lewis JC. Metal-responsive regulation of enzyme catalysis using genetically encoded chemical switches. Nat Commun. 2022 Apr 6;13(1):1864. doi: 10.1038/s41467-022-29239-y. PMID: 35387988; PMCID: PMC8987029.
10、Garabedian BM, Meadows CW, Mingardon F, Guenther JM, de Rond T, Abourjeily R, Lee TS. An automated workflow to screen alkene reductases using high-throughput thin layer chromatography. Biotechnol Biofuels. 2020 Nov 9;13(1):184. doi: 10.1186/s13068-020-01821-w. PMID: 33292503; PMCID: PMC7653764.
11、Burén S, Young EM, Sweeny EA, Lopez-Torrejón G, Veldhuizen M, Voigt CA, Rubio LM. Formation of Nitrogenase NifDK Tetramers in the Mitochondria of Saccharomyces cerevisiae. ACS Synth Biol. 2017 Jun 16;6(6):1043-1055. doi: 10.1021/acssynbio.6b00371. Epub 2017 Mar 3. PMID: 28221768; PMCID: PMC5477005.
12、Blount BA, Ellis T. The Synthetic Genome Summer Course. Synth Biol (Oxf). 2018 Nov 27;3(1):ysy020. doi: 10.1093/synbio/ysy020. PMID: 32995526; PMCID: PMC7445779.
13、Haines MC, Carling B, Marshall J, Shenshin VA, Baldwin GS, Freemont P, Storch M. basicsynbio and the BASIC SEVA collection: software and vectors for an established DNA assembly method. Synth Biol (Oxf). 2022 Oct 11;7(1):ysac023. doi: 10.1093/synbio/ysac023. PMID: 36381610; PMCID: PMC9664905.
14、Exley K, Reynolds CR, Suckling L, Chee SM, Tsipa A, Freemont PS, McClymont D, Kitney RI. Utilising datasheets for the informed automated design and build of a synthetic metabolic pathway. J Biol Eng. 2019 Jan 18;13:8. doi: 10.1186/s13036-019-0141-z. PMID: 30675181; PMCID: PMC6339355.
