题目:The gut microbiota promotes hepatic fatty acid desaturation and elongation in mice
多组学联合分析研究肠道微生物群促进小鼠肝脏脂肪酸去饱和和延长
期刊:Nature Communications
影响因子:12.353
研究背景
哺乳动物肠道具有高度复杂的肠道微生物群落,在过去的几十年中,许多小鼠模型已被用于研究肠道微生物群及其与宿主的关系。本研究整合转录组学、蛋白质组学、磷酸化蛋白质组学和脂质组学进行分析,提出了肠道微生物对肝脏脂质代谢的影响。
研究结果
1. 转录组和蛋白质组联合分析
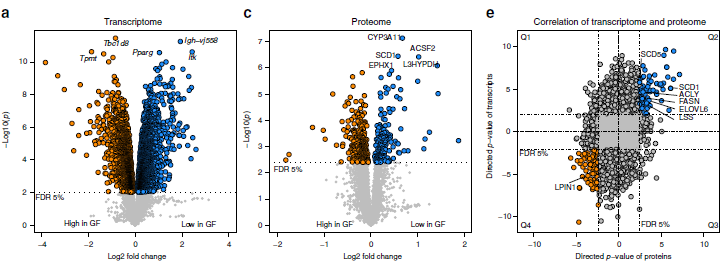
作者针对无菌小鼠模型(GF)和无特定病原体小鼠模型(SPF)的肝脏和血浆样品进行多组学分析。其中,小鼠肝脏样本的转录组使用微阵列芯片技术分析,总共分析了41174个探针,其中32308个可以定位到转录本,7769个探针显着差异表达(3937个基因),GF和SPF之间的差异倍数最大达到14.3(图1a)。通路富集分析揭示了GF小鼠中显著富集的多个代谢通路,如药物代谢、脂质代谢(图1b)。
使用TMT技术对小鼠肝脏样本进行蛋白质组学检测分析,共鉴定到5875个蛋白质,GF和SPF中存在显著差异的蛋白数目为455(图1c),差异倍数最高达3.7。差异表达最显著的蛋白质分别为L3HYPDH、CYP3A11、EPHX1、ACSF2、SCD1,这几个重要的蛋白质均参与了特定的代谢过程,如亚油酸和视黄醇代谢(图1d)。
作者使用基因名称合并了蛋白质组和转录组数据,以便识别在两个组学层中上调或下调的基因。两次组学同时鉴定到4843个基因(1i),其中2822个基因(约58%)为GF和SPF共有,984个基因在SPF中持续高表达(图1e,Q2),1,838个基因在GF中持续高表达(图1e,Q4)。蛋白质组和转录组基因功能富集分析显示SPF小鼠中上调的基因(Q2)在脂质代谢中参与的8个GO生物过程中有5个表现出富集(图1f),下调基因(Q4)则没有显著的GO注释。作者发现,影响脂质代谢的关键酶,如ACLY、FASN、SCD1和ELOVL6,在转录组与蛋白质组中均存在差异表达。
图1 SPF和GF小鼠肝脏样品的转录组学和蛋白质组学分析
2. 磷酸化蛋白质组分析
蛋白质磷酸化在酶的活化和失活中起重要作用,因此,作者同时对GF和SPF小鼠的肝脏样品进行磷酸化蛋白质组分析(磷酸化TMT)。在本次试验中,共鉴定到了5558种磷酸化肽段,其中1551个磷酸肽段,还可以检测相应的蛋白质,转录组、蛋白质组和磷酸化蛋白质组共同检测到1527个磷酸肽段(图1i)。作者研究发现363种蛋白质在GF和SPF动物之间具有显着不同的磷酸化水平(图1g),其中,8种蛋白参与到了脂质代谢ACOT1、FASN、GPAM、LPIN1 / 2、PGRMC2、PLIN3和SLCO1A4。
3. 脂质组学分析
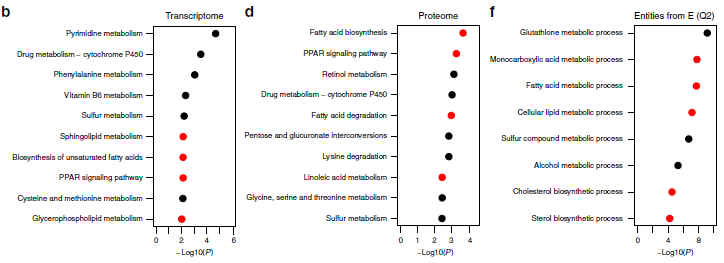
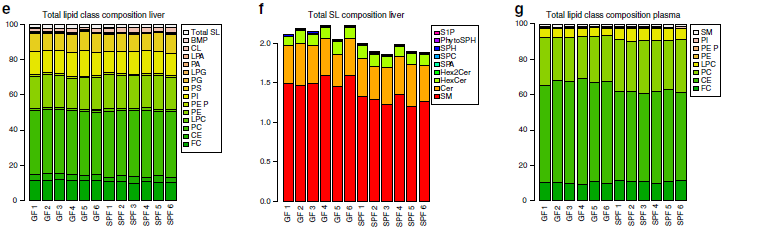
为了检测脂肪酸和脂质代谢中涉及的酶的变化是否导致GF小鼠中的脂质水平改变,作者进行了全面的定量脂质组分析。使用气相色谱-质谱联用(GC-MS)技术共检测到血浆与肝脏样本中的20类525种脂质,如脂肪酸(FAs)、甘油磷脂、鞘脂和甾醇等。作者研究发现肝脏样本中的16种脂质在GF和SPF小鼠之间差异显著,血浆样本中则15种脂质差异显著。最重要的是,作者发现SPF和GF小鼠之间存在从单不饱和脂质向多不饱和脂质系统转变的趋势。SPF小鼠样品中的棕榈油酸和含有单不饱和酰基链的甘油磷脂,含量高出1.5倍(图2a-d),而在GF小鼠中,多不饱和脂肪酸和甘油磷脂(如花生四烯酸)的含量较高。磷脂酰胆碱(PC,37%)、磷脂酰乙醇胺(PE,19%)、游离胆固醇(FC,11%)是肝脏中的主要脂质类别(图2e),鞘脂仅代表肝脏脂质的一小部分,约占2%(图2f)。血浆脂质则以磷脂酰胆碱(PC,27%)、胆固醇酯(CE,54%)和游离胆固醇(FC,10%)为主(图2g),而且肝脏和血浆样品中每种脂质类别的总浓度没有差异(图2e,g)。
总而言之,作者认为以上数据显示肠道微生物群影响肝脏和血浆中的特定脂肪酸和甘油磷脂物种谱。含有肠道微生物群的小鼠会较高比例的含有单不饱和脂肪酸(MUFA),而GF小鼠以脂质饱和脂肪酸(SAFA) 和多不饱和脂肪酸(PUFA)为主。
图2 GF和SPF小鼠肝脏和血浆的定量脂质组分析
4. 组学数据重建脂肪酸代谢
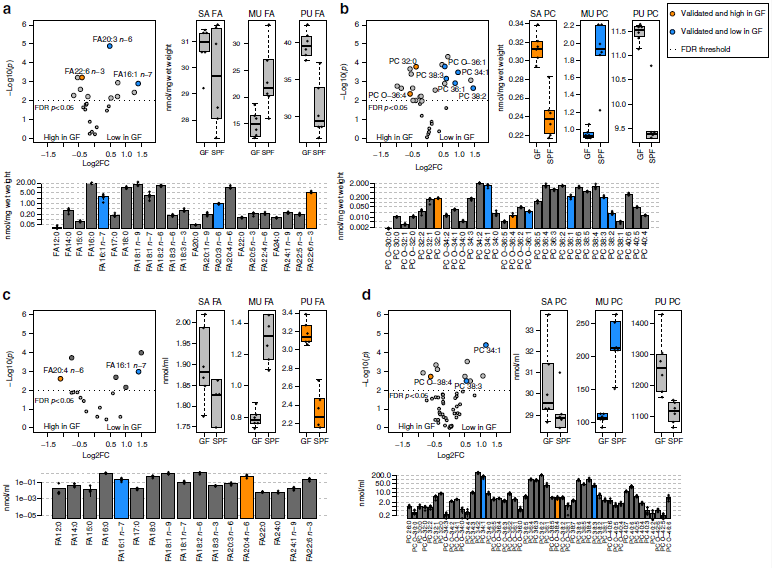
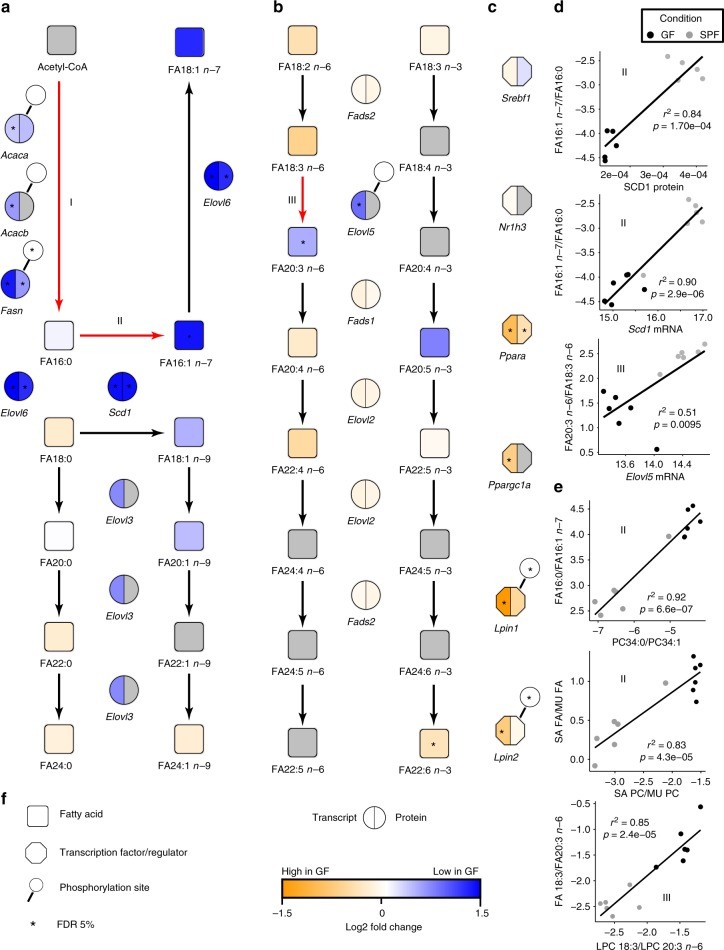
作者假设GF小鼠的脂肪酸饱和度和延伸能力与SPF小鼠不同,导致肝脏甘油磷脂酰基链谱改变。因此,作者重建了脂肪酸代谢途径并整合了磷酸化蛋白质组、蛋白质组和转录组,系统地概述肝脏脂质代谢过程(图3a,b)。作者在3个特定的脂肪酸转化步骤中观察到GF和SPF小鼠之间的显著差异:(I)借助ACAC A / B和FASN,从乙酰-CoA从头合成脂肪酸的过程(图3a); (II)通过SCD1将棕榈酸酯(FA16:0)去饱和为棕榈油酸酯(FA16:1 n-7)(图3a); (III)通过ELOVL5(图4b)将FA18:3 n-6延伸至FA20:3 n-6(图3b)。在所有步骤中,SPF具有较高值。为了支持(II)和(III),作者将脂肪酸产物/前体比率与相应的mRNA或蛋白质丰度相关联。如图 3d所示 ,SCD1表达与16:1 n -7 / FA16:0比率(mRNA: R = 0.93; p <0.00002;蛋白质:R = 0.94; p <0.00005)强相关,ELOVL5表达与20:3 n -6/18:3 n -6的比率(mRNA: R = 0.79; p <0.002; 蛋白质:未检测到)强相关。此外,作者还发现脂肪酸部分的反应比(产物/前体水平)与PC和LPC物种的相应去饱和度和伸长指数高度相关(图3e),表明改变的FA代谢过程清楚地反映在酰基中。
图3 多组学数据重建脂肪酸代谢途径
5. 基于脂肪酸谱的综合分类
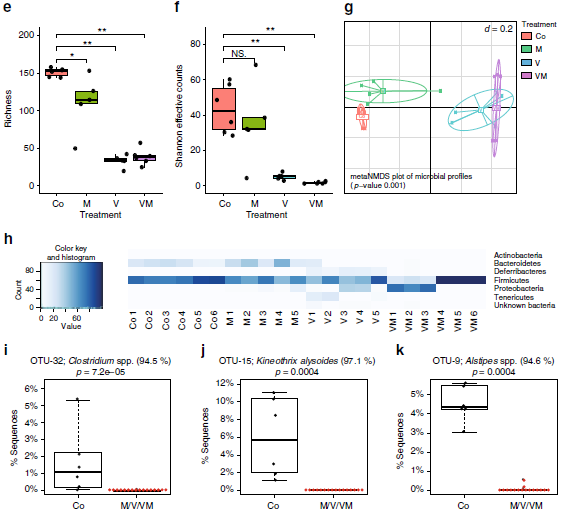
为了获得肠道微生物群特异性调节脂质代谢过程的进一步证据,作者利用短期抗生素(氨苄青霉素(A)、万古霉素(V)、甲硝唑(M)、或万古霉素(V)和甲硝唑(M)的组合)处理SPF的肠道微生物生态系统,GC-MS定量肝脏脂肪酸谱,作者发现通过FA 16:0、FA 20:3 n-6、FA 20:4 n-6和FA 22:6 n-3进行分类,既能清楚地将GF与SPF小鼠区分开(图4a,b),也可将不同抗生素处理与未处理的小鼠区分开(图4c)。Scd1和Elovl5的mRNA表达水平明显遵循分类评分的趋势,低评分样本有低表达水平(图4d)。这些结果支持了肠道微生物群影响脂肪酸代谢代谢,特别是由SCD1和ELOVL5产生的脂肪酸(图3a,b;反应II和III)。
图4抗生素对肠道微生物种类的影响
6. 肠道微生物群组成分析
为了确定抗生素对肠道微生物群多样性和组成的影响,作者通过高通量测序分析了16S rRNA基因的V3-V4扩增子。本次实验共检测总到376071个序列,代表172个OTU(每个样品99±49 OTU)。所有抗生素均影响α多样性(图4e,f),与单独的M相比,V和VM的组合具有特别显著的影响,β多样性分析揭示了V和VM样品的显著聚集,这些样品与对照组分开,但是彼此并不分离(图4g),受抗生素影响最大的OUT来自于厚壁菌和拟杆菌。
文章小结
宿主的肠道微生物与脂质稳态之间的相互作用,与宿主的生理学机能和代谢疾病高度相关。本研究整合转录组学、蛋白质组学、磷酸化蛋白质组学和脂质组学进行分析,提出了肠道微生物促进肝脏脂质代谢的多组学观点。微生物可以通过硬脂酰辅酶A脱氢酶1(SCD1)诱导单不饱和脂肪酸生成,并通过脂肪酸延长酶5诱导多不饱和脂肪酸延伸,导致甘油磷脂酰基链分布的显著改变。
解析文献
Alida Kindt, Gerhard Liebisch, et al. The gut microbiota promotes hepatic fatty acid desaturation and elongation in mice. Nature Communications, 2018, DOI: 10.1038/s41467-018-05767-4
参考文献
1. Rappsilber, J., Mann, M. & Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics usingStageTips. Nat. Protoc, 2007, 2, 1896–1906.
2. Ruprecht, B. et al. Evaluation of kinase activity profiling using chemical proteomics. ACS Chem. Biol. 2015, 10, 2743–2752.
3. Bartram, J. et al. Accurate sample assignment in a multiplexed, ultrasensitive, high-throughput sequencing assay for minimal residual disease. J. Mol. Diagn.2016, 18, 494–506.
4. Ruprecht, B., Zecha, J., Zolg, D. P. & Kuster, B. High pH reversed-phase microcolumns for simple, sensitive, and efficient fractionation of proteome and (TMT labeled) phosphoproteome digests. Methods Mol. Biol. 2017, 1550, 83–98.