基本信息
货号:13601
储存条件:保存在冰箱中,避光
适用仪器:荧光显微镜
简介
组蛋白去乙酰化酶(HDAC)是一类从组蛋白上的ε-N-乙酰基赖氨酸氨基酸中除去乙酰基的酶。去乙酰化恢复赖氨酸氨基酸的正电荷,这增加了组蛋白对DNA的带负电荷的磷酸骨架的亲和力。该过程通常通过阻断转录因子的进入来下调DNA转录。正在研究HDAC抑制剂作为癌症的治疗方法。
我们的Amplite™荧光HDAC活性检测试剂盒为检测HDAC活性提供了一种快速,方便,灵敏的方法。该试剂盒使用我们的非肽HDAC Green™底物,它比基于肽的HDAC底物(如Ac-RGK(Ac)-R110,Ac-RGK(Ac)-AMC和Ac-RGK(Ac) - 更敏感 - AFC。此外,HDAC Green™底物比其他商业肽基HDAC底物更耐蛋白酶水解。我们的试剂盒可用于测量细胞裂解液中的HDAC活性或用细胞提取物或纯化酶筛选HDAC抑制剂。 HDAC Green™底物的长波长发射使得该测定不受化合物和细胞组分的干扰。用490nm的激发和525nm的发射监测HDAC活性。

试剂盒主要特点
广泛应用:可用于量化溶液和细胞提取物中的HDAC。
连续性:无需分离步骤即可轻松适应自动化。
方便:配方具有最小的手动操作时间。 不需要洗涤。
非放射性:对废物处理没有特殊要求。
试剂盒组成

96孔板样品测定方案
简要概括
准备含有HDAC的样品(40μL)®
添加HDAC抑制剂或测试化合物(10μL)®
在室温或37 oC孵育10-20分钟®添加HDAC Green™底物工作溶液(50μL)®
在室温下孵育或 37 oC持续30-60分钟®
在Ex / Em = 490/525 nm处监测荧光强度
操作步骤
1.准备工作解决方案:
1.1制备含有HDAC的测试样品:在测定缓冲液(组分B)中以1:40稀释5-10mg / mL HeLa核提取物或细胞裂解物。
注意:40μL稀释样品足以用于96孔板的一个孔。使用前立即稀释提取物。将溶液储存在冰上。
1.2制备HDAC抑制剂(Trichostain A)溶液的稀释液:在测定缓冲液(组分B)中以1:100稀释3mM曲古抑菌素A溶液(组分C),得到30μM曲古抑菌素A溶液。向每个抑制剂对照孔中加入10μL30μM曲古抑菌素A溶液。
1.3准备HDAC Green™底物工作溶液:将20μLHDACGreen™底物(组分A)和100μL信号增强剂(组分D)加入5 mL分析缓冲液(组分B)中。
注意1:稀释的HDAC Green™底物工作溶液不稳定,5 mL稀释的HDAC Green™底物工作溶液足以进行100次分析。
注2:为每个实验准备新鲜的HDAC Green™底物工作溶液。将重构的工作溶液保持在冰上直至使用。
2.运行HDAC分析:
2.1将40μL稀释的核提取物,酶溶液或其他HDAC样品和10μL测试化合物加入相应的微孔板孔中(见表1)。
对于阳性对照:加入40μL稀释的HDAC酶溶液或HeLa核提取物(来自步骤1.1)和10μL分析缓冲液(组分B)。对于阴性对照:加入40μL稀释的HeLa核提取物(来自步骤1.1),加入10μL μL的30μM曲古抑菌素A溶液(来自步骤1.2),或使用不含HDAC活性的已知样品。对于空白(无酶):仅添加50μL的测定缓冲液(组分B)。
2.2在室温或37℃孵育平板10-20分钟。
注意:为了筛选HDAC抑制剂,在添加HDAC Green™底物工作溶液之前,先用HeLa核提取物或纯酶预孵育这些化合物(参见步骤2.3)
2.3将50μLHDACGreen™底物工作溶液(来自步骤1.3)加入每个孔中。将板在室温或37℃孵育30-60分钟。
2.4在Ex / Em = 490/525 nm处监测荧光强度。
表1.在96孔微孔板中的测试化合物的核提取物的布局

数据分析
空白孔中的荧光(仅使用测定缓冲液)用作背景荧光,并且用具有HDAC Green TM反应的那些孔的值减去。 所有荧光读数均以相对荧光单位(RFU)表示。 典型数据如图1所示。
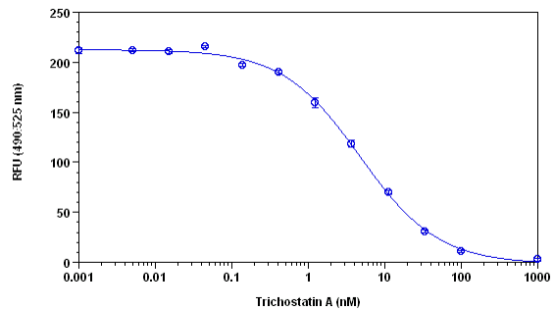
图1.曲古抑菌素A使用Gemini荧光酶标仪(Molecular Devices),用Amplite TM荧光HDAC活性测定试剂盒测量HeLa核提取物中的抑制。
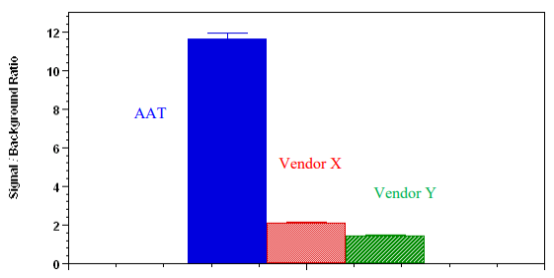
图2.使用Amplite™荧光测定法测定HeLa核提取物中的HDAC活性HDAC活性测定试剂盒(蓝色)与供应商X(红色)和供应商Y(绿色)进行比较,两者均使用Ac-RGK(Ac) -R110肽底物。 使用Amplite™荧光HDAC活性测定试剂盒测量的HDAC活性的信号/背景比率比供应商X和Y高10倍以上。
参考文献
1. Belien A, De Schepper S, Floren W, Janssens B, Marien A, King P, Van Dun J, Andries L, Voeten J, Bijnens L, Janicot M, Arts J. (2006) Real-time gene expression analysis in human xenografts for evaluation of histone deacetylase inhibitors. Mol Cancer Ther, 5, 2317.
2. Takahashi-Fujigasaki J, Fujigasaki H. (2006) Histone deacetylase (HDAC) 4 involvement in both Lewy and Marinesco bodies. Neuropathol Appl Neurobiol, 32, 562.
3. Olaharski AJ, Ji Z, Woo JY, Lim S, Hubbard AE, Zhang L, Smith MT. (2006) The histone deacetylase inhibitor trichostatin a has genotoxic effects in human lymphoblasts in vitro. Toxicol Sci, 93, 341.
4. Voelter-Mahlknecht S, Ho AD, Mahlknecht U. (2005) FISH-mapping and genomic organization of the NADdependent histone deacetylase gene, Sirtuin 2 (Sirt2). Int J Oncol, 27, 1187.
5. Klampfer L, Huang J, Swaby LA, Augenlicht L. (2004) Requirement of histone deacetylase activity for signaling by STAT1. J Biol Chem, 279, 30358.
6. Nusinzon I, Horvath CM. (2003) Interferon-stimulated transcription and innate antiviral immunity require deacetylase activity and histone deacetylase 1. Proc Natl Acad Sci USA, 100, 14742.
