BSA介绍
集群分离分析(Bulk Segregant Analysis,BSA),即利用亲本与极端性状个体构建的混池,挖掘与性状相关的突变位点信息,后续可作为分子标记辅助分子育种。常用的性状定位分析方法为SNP-index方法。
BSA优势总结
1、与传统定位方法比较,大大降低了人力与时间成本,快速获得性状定位结果;
2、只需要检测亲本与极端个体混池,即可获得性状关联的突变位点信息,降低了研究成本;
3、不受亲本限制,多种群体均可进行性状定位研究;
4、通过circos图,可视化性状定位结果,更直观展示;
5、博奥晶典专门针对多倍体与基因组复杂的物种推出全新的BSA分析策略,完美解决多倍体或复杂基因组物种性状定位难题。
图1:SNP-index计算方法
注:SNP-index 表示突变基因型的覆盖度/此位点总覆盖度,在第一个突变位点,相比较参考基因型,突变基因型为G,覆盖度为4,而此位点覆盖度为10,SNP-index=4/10=0.4。
方案建议
1、群体类型:F1、 F2、DH 、RIL等群体;
2、样本选择:
• 双亲本+2个F2极端性状子代混池
• 双亲本+1个混池
• 1个野生亲本+1个突变混池(EMS诱变个体与野生型构建的群体)
• 单亲本+1个混池
• 无亲本+2个极端混池(F2,F1等)
3、适用样本参考
A. 针对所有有参考基因组物种;
B. 多倍体物种:如小麦等;
C. 基因组重复序列含量高的物种:如玉米等;
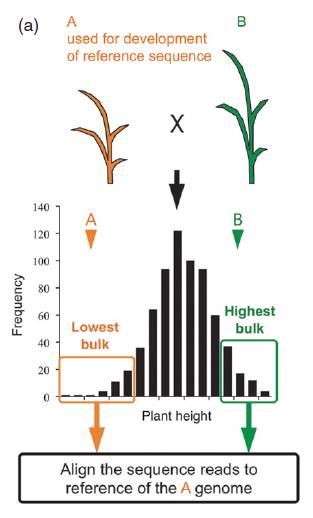
图2:QTL-seq群体选择示意图
注:QTL-seq适用于数量性状的群体,选择表型极端的个体构建混池。
结果展示
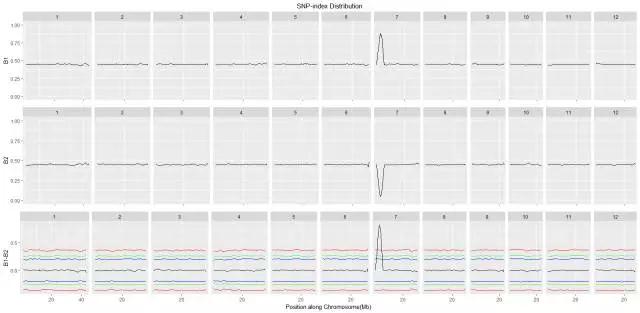
SNP-index分布示意图:
下图分为上中下三个部分,横坐标表示各染色体,上图表示混池B1的SNP-index值在各染色体上的分布,中间的图表示混池B2的SNP-index值在各染色体上的分布,下图黑线表示两个混池的SNP-index差值,即ΔSNP-index,在各染色体上的分布,蓝绿红线分别表示不同的置信水平,从上到下依次为0.99,0.95,0.9,0.1,0.05,0.01的置信水平,超过置信水平的区域为性状相关的候选区域。
图3:SNP-index分布示意图
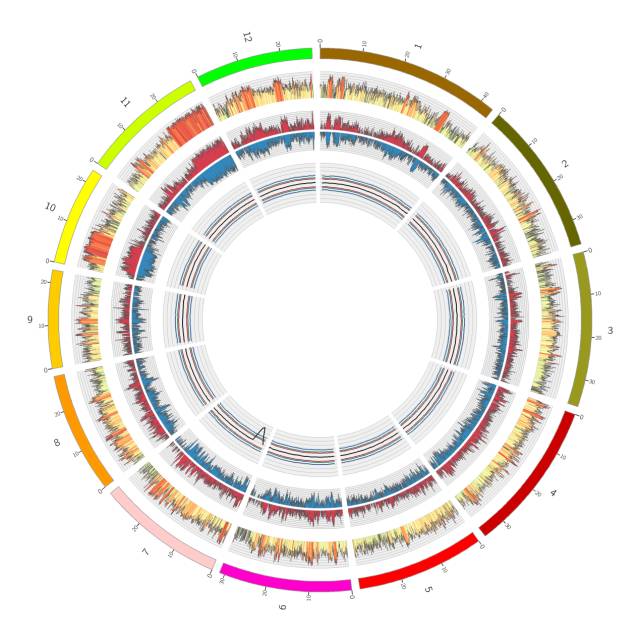
下图从外到内,第一圈表示参考基因组的染色体;第二圈表示SNP的密度分布,柱状颜色越红越高表示密度越高,越蓝越低表示密度越低;第三圈红色表示Insert的密度分布,柱越高表示密度越高;第四圈蓝色表示Delete的密度分布,柱越高表示密度越高;第五圈表示ΔSNP-index分布,黑线表示平均ΔSNP-index,蓝线,绿线,红线分别表示0.01,0.05,0.1,0.9,0.95,0.99的置信水平。
图4:Circos可视化定位结果示意图
参考文献
[1] Abe A, KosugiS, Yoshida K, et al. Genome sequencing reveals agronomically important loci inrice using MutMap[J]. Nature Biotechnology, 2012, 30(2):174-8.
[2] Takagi H, Abe A,Yoshida K, et al. QTL‐seq: rapid mapping of quantitative trait loci in riceby wholegenome resequencing of DNA from two bulked populations[J]. Plant Journal,2013,74(1):174-83.
产品详情:http://www.capitalbiotech.com/ProductServer.html?categoryId=50
撰稿 | 博奥晶典生物信息部 樊淑
文案 | 博奥晶典科研服务事业部 李莹、吴洁、张西轩
部分配图来源于网络,侵删
微生物组与代谢组联合研究 | 母婴健康相关的研究和应用专题
(2020-12-17T15:38 浏览数:18934)
会议邀约 | 科研领域最热技术,单细胞组学技术与应用研讨会,正式开启!
(2020-09-23T10:04 浏览数:19133)
微生物组与代谢组联合研究 | 药物领域的研究和应用专题
(2020-08-31T16:43 浏览数:18614)
项目文章|博奥晶典单细胞测序平台支持发文研究新冠肺炎患者的适应性免疫反应
(2020-08-27T17:27 浏览数:19224)
项目文章 | 博奥晶典转录组测序支持发文——疾病研究篇
(2020-08-19T15:04 浏览数:18464)
Nat Immunol | 博奥晶典单细胞项目文章揭示新冠肺炎发病过程中整体免疫反应特征及相关规律
(2020-08-17T17:26 浏览数:17588)
4步教你快速get单细胞数据分析套路
(2020-08-12T16:16 浏览数:18108)
暑期福利 | 植物原生质体制备大揭秘!
(2020-08-07T10:19 浏览数:17772)
解密 | 消化处理几千个样本后总结的单细胞样本处理注意事项
(2020-07-28T09:20 浏览数:18363)
新品发布 | 博奥晶典单细胞靶向基因检测产品接受预定
(2020-08-12T16:16 浏览数:18535)