乳腺癌是女性中最常见的癌症之一,是导致女性癌症致死的主要原因。这其中又以后期骨转移导致的骨组织癌症居多,因此研究乳腺癌相关的癌细胞转移机制就显得尤为重要。在癌细胞转移性播散过程中,来自原发肿瘤的乳腺癌细胞必须首先进行上皮-间充质转化侵染周围组织,进入血液循环系统的微血管,进而转移到骨组织内。RUNX2 (Runt-related transcription factor 2, RUNX2)是一种与成骨相关的转录因子,已经成为一个重点研究的致癌过程中的转录抑制因子。然而,RUNX2在乳腺癌转移中的作用尚不明确。
2022年5月9日,黄蔚课题组和王艳课题组在Cell Death & Differentiation上发表题为RUNX2 recruits the NuRD(MTA1)/CRL4B complex to promote breast cancer progression and bone metastasis的研究成果。
该研究证实了RUNX2促进并结合 metastasis-associated 1 (MTA1)/NuRD和Cullin 4B (CUL4B)-Ring E3 ligase (CRL4B)复合物形成转录抑制复合物,催化组蛋白去乙酰化和泛素化。对RUNX2/NuRD(MTA1)/CRL4B复合靶点的全基因组分析确定了包括过氧化物酶体在内的一组基因增殖因子:激活受体α (PPARα)和超氧化物歧化酶2 (SOD2)在细胞生长,上皮-间充质转化(EMT)和细胞侵袭中起重要作用。
该研究证明了RUNX2/NuRD(MTA1)/CRL4B复合物促进体外和体内乳腺癌的增殖、侵袭、肿瘤发生、骨转移、癌干性,表明RUNX2在将来乳腺癌治疗发展中可以作为潜在靶点。

该研究首先通过数据库分析得出RUNX2上调与乳腺癌发生相关,并且进一步的实验结果表明RUNX2调节肿瘤抑制基因的表达和参与骨转移相关信号通路。
实验结果显示了几个最常见的肿瘤抑制因子在RUNX2被敲减后,基因表达上调,包括BAX, EIF3F, FADD, PPARα,FBXW7, HSP90B1, CASP 7, SIAH2, TNFAIP3,TSC22D1和SOD2。此外,还有几个癌相关基因在RUNX2被敲减后的细胞中出现下调。这些结果显示RUNX2可能与乳腺癌的发生有关。
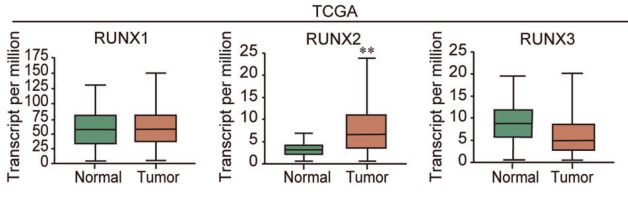
图1. RUNX家族在正常及乳腺癌组织中基于GEO和TCGA数据库的表达分析,结果表明RUNX2在癌细胞的转录频率差异相较于正常细胞最高。
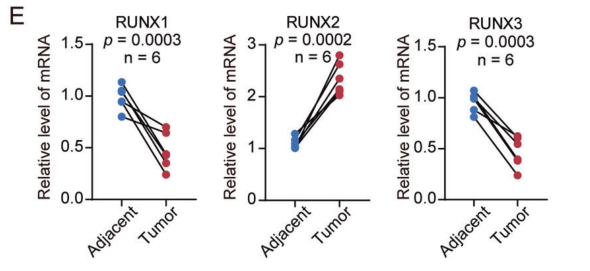
图2. RUNX2在乳腺癌组织中的表达量显著高于癌旁组织中的表达。
免疫亲和纯化和质谱分析结果表明RUNX2与NuRD(MTA1)和CRL4B复合物相关联,进一步的纯化结果表明NuRD (MTA1)和CRL4B复合体与RUNX2可以直接结合互作。
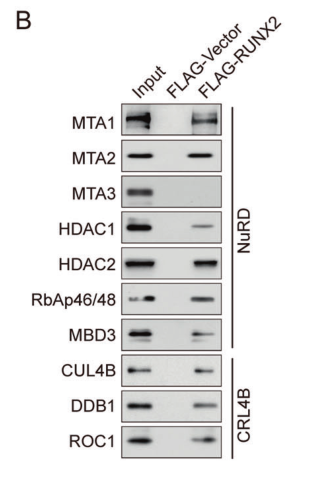
图3. 使用MDA-MB-231细胞FLAG抗体对纯化的部分进行Western blotting分析,结果表明NuRD(MTA1)和CRL4B的组分含量较高。
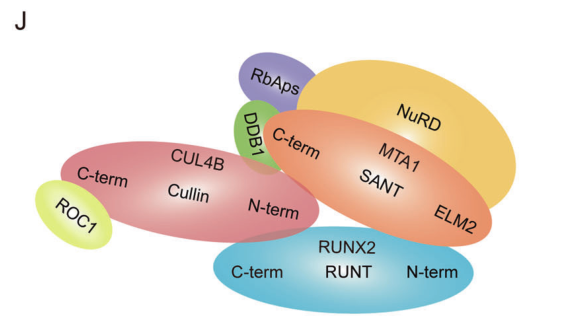
图4. RUNX2/NuRD(MTA1)/CRL4B复合体分子相互作用示意图
细胞实验结果表明 RUNX2、MTA1或CUL4B的敲减导致上皮间质转化相关标记因子 fibronectin、N-cadherin和vimentin的表达减少,并诱导间充质标记因子E-cadherin、α-catenin和γ-catenin的表达,而RUNX2、MTA1或CUL4B的过表达得到的结果刚好相反。
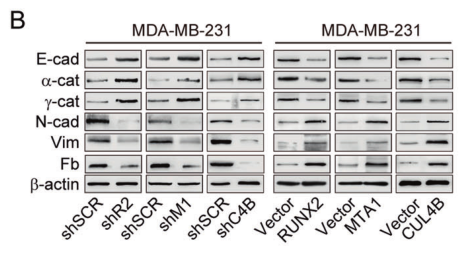
图5. MDA-MB-231细胞中RUNX2、MTA1或CUL4B敲减或过表达后相应上皮和间充质标记因子的表达量变化。
另外干细胞标志物在RUNX2、MTA1或CUL4B敲减后下调,在RUNX2、MTA1或CUL4B正常表达时上调,以及 细胞成球实验和肿瘤生长实验的结果都说明RUNX2/NuRD(MTA1)/CRL4B可以促进癌细胞增殖。
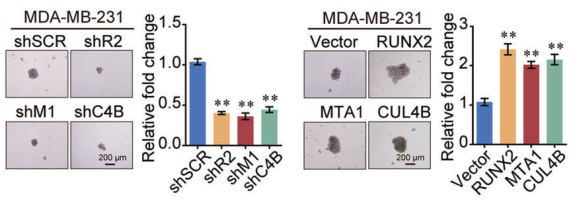
图6. 细胞成球实验结果表明RUNX2、NuRD(MTA1)和CRL4B都可以促进癌细胞增殖。
RNA-seq 和KEGG分析结果表明,RUNX2和MTA1共同参与PI3K-AKT, HIF-1和 IL-17信号通路。qChIP分析显示RUNX2/NuRD(MTA1)/CRL4B复合物大量富集肿瘤抑制基因启动子(SOD2、CASP7、PPARα、BAX、SIAH2、ANXAN7、FBAW7、EIF3F、EI24、TSC22D1、EGR1和NEURL1)。
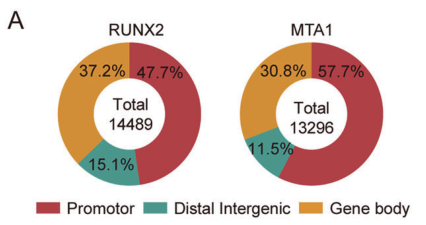
图7. 使用CUT&Tag技术分析确定RUNX2和MTA1的基因组分布。
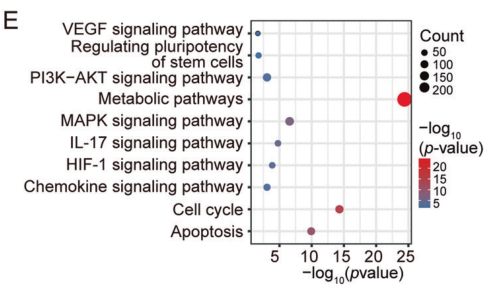
图 8. 3857个与RUNX2/MTA1/CUL4B复合物结合的启动子(重叠区域)基因的KEGG通路分析
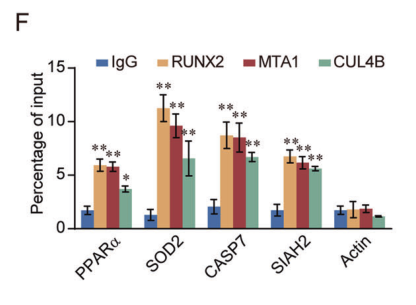
图9. qChIP结果表明SOD2、CASP7、PPARα 、SIAH2的基因启动子和RUNX2、NuRD(MTA1)和CRL4B的富集度均较高。
进一步的qChIP实验表明,RUNX2、MTA1或CUL4B的敲减导致目标基因启动子上相应蛋白的募集显著减少。另外,在WB实验中也得到了类似结果。综上所述,这些结果证实了RUNX2、NuRD(MTA1)和CRL4B通过功能性结合来对多组抑癌靶基因(如PPARα、SOD2、CASP7和SIAH2)的转录进行抑制。
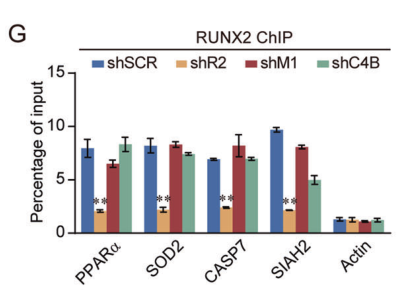
图10. qChIP结果表明SOD2、CASP7、PPARα 、SIAH2的基因启动子丰度在RUNX2敲减后均有显著降低。
该部分实验中应用了近岸蛋白的NovoNGS® CUT&Tag 3.0 High-Sensitivity Kit (for Illumina®)(Cat.No.:N259-YH01),首先使用CUT&Tag试剂盒对细胞样本依次进行ConA磁珠结合、一抗和二抗结合、 pAG-Tn5结合、 加入Tagmentation buffer片段化DNA,DN**段纯化后用于文库构建和高通量测序。接着,通过qChIP实验分析了RUNX2、MTA1或CUL4的结合位点,并成功确定了肿瘤抑制基因的富集启动子。后续用qChIP实验验证了RUNX2、MTA1或CUL4敲减对启动子富集的影响。
该研究发现RUNX2的敲减促进了上皮标记因子的表达,降低了EMT标记因子的表达,而共同敲除PPARα或 SOD2可以部分恢复该表型,这表明RUNX2可以通过抑制PPARα或 SOD2的表达促进乳腺癌细胞的侵袭,同时,骨转移相关标记因子表达的降低可以通过PPARα的共同敲减而得到恢复。这些结果表明RUNX2通过抑制PPARα和SOD2的表达促进乳腺癌细胞EMT和骨转移。
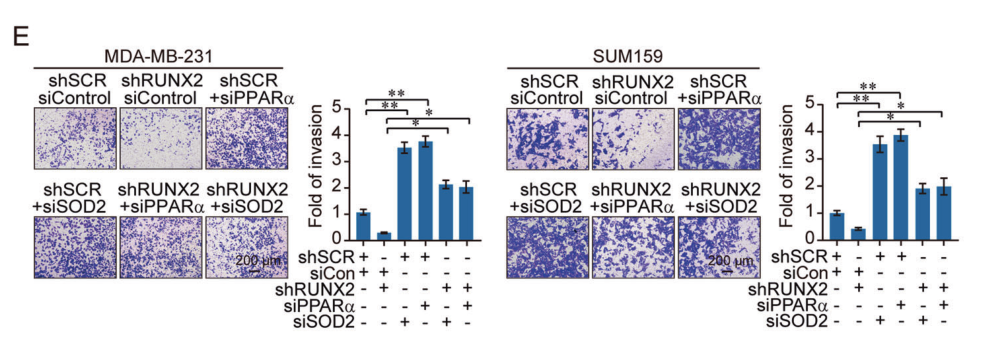
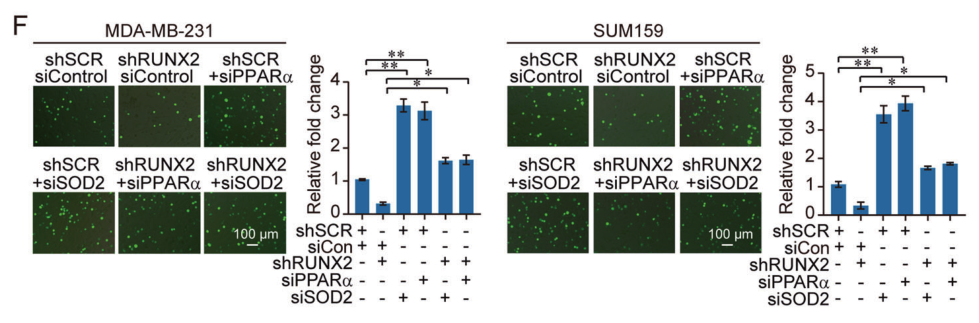
图11和12. MDA-MB-231和SUM159细胞的趋化迁移试验和癌细胞-骨基质粘附试验都表明RUNX2通过抑制PPARα和SOD2的表达促进乳腺癌EMT和骨转移。
综上所述,RUNX2在包括乳腺癌在内的多种癌症中表达上调,而PPARα作为RUNX2的下游调节因子在乳腺癌中表达下调,提示RUNX2可能作为乳腺癌的生物标记因子。RUNX2可以与NuRD(MTA1)/CRL4B复合体合作,并且诱发癌细胞增殖、侵袭、骨转移和癌干细胞特性。此外,RUNX2/NuRD(MTA1)/CRL4B复合体有助于肿瘤抑制因子的表观遗传沉默。PPARα和SOD2被证实是RUNX2/NuRD(MTA1)/CRL4B复合体的靶基因。本研究为RUNX2在肿瘤发生和转移中的作用提供了新的转录调控模型和新的分子基础,表明RUNX2是一种潜在的治疗手段癌症治疗的目标。
在该项研究中使用的试剂盒NovoNGS® CUT&Tag 3.0 High-Sensitivity Kit (for Illumina®)(Cat.No.:N259-YH01)来自近岸蛋白。产品引用文献已有10余篇,为研究蛋白与DNA互作提供了帮助,为整个研究奠定了基础。
苏州近岸蛋白质科技股份有限公司,是一家专注于重组蛋白应用解决方案的高新技术企业,主营业务为靶点及细胞因子类蛋白、重组抗体、酶及试剂的研发、生产和销售,并提供相关技术服务。公司定位为医疗健康与生命科学领域原料与技术解决方案的上游供应商,致力于为下游客户提供及时、稳定、优质的产品及服务,助力全球生物医药企业和研究机构的技术与产品创新升级。
详询www.novoprotein.com.cn或致电400-600-0940。
